
近日,深圳湾实验室周耀旗课题组、詹剑研究员、北大深圳研究生院及鹏城实验室陈杰课题组和现在上海人工智能实验室的高志强研究员合作在Nucleic Acids Research上发表研究文章:“Multiple sequence-alignment-based RNA language model and its application to structural inference”(基于多序列比对RNA语言模型及其在结构推衍中的应用),为语言模型在RNA结构预测上的应用打响了第一枪。
最近语言模型ChatGPT在回答各类问题的范围、深度和准确度上的革命性进展,掀起了开发通用大语言模型的热潮。与此同时,这些语言模型在生物高分子蛋白质上的应用也在不断深入,已经有二十多个不同规模、不同应用的模型问世,在蛋白质结构和功能预测、以及蛋白质设计上发挥出越来越大的作用。与有20个氨基酸组成的蛋白质相比,RNA语言模型的开发更为困难,因为RNA仅由4种碱基的排列组成,序列保守性极差,从而利用无监督训练来捕捉其中的结构和功能信息的挑战极大。
近日,深圳湾实验室周耀旗课题组、詹剑研究员与北大深圳研究生院及鹏城实验室陈杰课题组合作在Nucleic Acids Research上发表研究文章:“Multiple sequence-alignment-based RNA language model and its application to structural inference”(基于多序列比对RNA语言模型及其在结构推衍中的应用),成功开发了一个无监督多序列比对的RNA语言模型【1】。
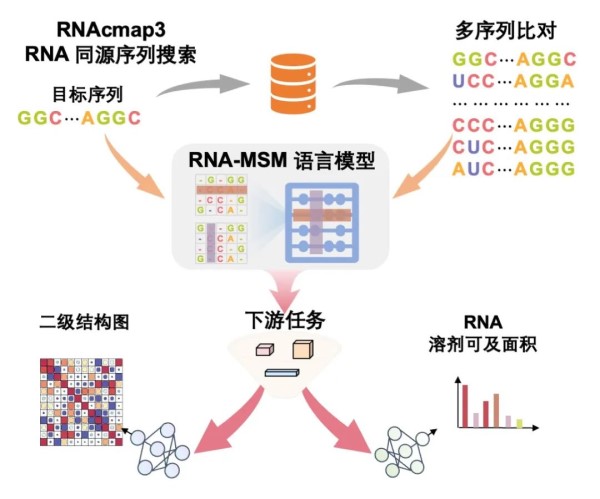
据了解,他们决定开发基于同源序列比对、而不是基于单个序列的语言模型,是因为发现基于BERT(Bidirectional Encoder Representations from Transformers,双向编码器变换器)的RNA语言模型,无论是内部开发的,还是2022年在预印版arxiv上公开的,对三级结构包含的RNA碱基对的预测没有带来任何明显的效果。因此,提供多序列比对,然后让语言模型去提取进化和共进化信息可能会更加有效。但是,被认为是金标准的、人工收集标注的RNA家族(Rfam)里面只有4000个家族,而且每个家族里的RNA同源序列数的中位数只有45个,远远无法满足深度学习的需求。所以,他们不得不利用自主研发的全自动RNAcmap3管线,来从自己构建的、目前最大的核酸数据库MARS中,利用RNAfold预测的近似二级结构、并通过Infernal来搜索具有相同二级结构的同源序列。这个搜索提供了远超Rfam的同源序列数目(中位数>2000)。研究发现,这个多序列比对语言模型RNA-MSM所产生的二维注意力图和一维嵌入层已经自动包含了结构信息,可以直接映射到RNA的碱基配对概率和溶剂可及性。通过进一步的微调,这一模型在RNA结构相关任务上表现出了大幅度的改善,超过了现有的技术,包括利用了进化信息的SPOT-RNA2【2】和RNAsnap2【3】。
这一进展意味着RNA领域的结构推衍有了新的工具和方法,有望在RNA的结构和功能相关任务中发挥重要作用。团队将继续致力于研究和改进这一RNA语言模型,以进一步提高其通用性和泛化能力,为RNA的三维结构预测提供更多可能性。另一方面,该发现有望加速我们对RNA分子的理解,对生命科学研究和医学应用产生深远影响。团队将继续不懈努力,以更好地理解RNA的结构和功能,为新药研发和疾病治疗带来更多可能。
该工作得到国家自然科学基金委和深圳市科创委专项资金的支持。
参考文献
【1】Y. Zhang, M. Lang, J. Jiang, Z. Gao, F. Xu, T. Litfin, K. Chen, J. Singh, X. Huang, G. Song, Y. Tian, J. Zhan, J. Chen, and Y. Zhou, “Multiple sequence-alignment-based RNA language model and its application to structural inference.”, Nucleic Acids Research, in press (2023). https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkad1031/7369930?utm_source=authortollfreelink&utm_campaign=nar&utm_medium=email&guestAccessKey=0ea85dab-dfdc-4855-bc61-46a1076f738d
【2】J. Singh, K. Paliwal, T. Zhang, J. Singh, T. Litfin, and Y. Zhou, Improved RNA secondary structure and tertiary base-pairing prediction using evolutionary profile, mutational coupling and two-dimensional transfer learning, Bioinformatics, 37, 2589–2600 (2021). https://academic.oup.com/bioinformatics/article/37/17/2589/6168141?login=false
【3】 A. Kumar, J. Singh, K. Paliwal, J. Singh, Y. Zhou, “Single-sequence and profile-based prediction of RNA solvent accessibility using dilated convolution neural network.”, Bioinformatics, 36: 5169-5176 (2020). https://academic.oup.com/bioinformatics/article/36/21/5169/5873586?login=false
原文信息:
Multiple sequence-alignment-based RNA language model and its application to structural inference
课题组主页:
http://zhouyq-lab.szbl.ac.cn
来源 | 周耀旗课题组
编辑 | 鲍 啦
责编 | 远 山
欢迎投稿、建议 | media@szbl.ac.cn

 粤公网安备44031102000926号
粤公网安备44031102000926号