近期,深圳湾实验室系统与物理生物学研究所李刚课题组在Nature Communications期刊发表了题为“Spatiotemporal-resolved protein networks profiling with photoactivation dependent proximity labeling”的研究论文,论文开发了一种基于单线态氧邻近标记、且具有高时空分辨率的蛋白质互作网络分析的蛋白质组学平台。
蛋白质相互作用网络是细胞过程的基础,也是生物学最重要的研究工具之一。高时空分辨率地揭示蛋白相互作用图谱将为破译生物作用通路、解析病理学分子机制以及疾病治疗发挥重要作用。目前广泛采用的邻近标记方法,例如BioID / TurboID或者APEX等方法,虽然被研究人员大量采用,但均存在一定的局限性。例如TurboID方法利用“活化酯”作为标记中间体,其半衰期为分钟级,反应活性较差,导致标记背景较高,且存在一定的内源性生物素产生的背景;而APEX方法需要利用双氧水处理细胞,催化生成生物素-苯酚自由基,尽管其半衰期提高到毫秒级[1],但是其依然存在标记背景高的问题,且双氧水处理会影响对氧化敏感的蛋白或者细胞通路,并会对细胞产生一定的毒性。
因此作者认为发展新型邻近标记方法的关键在于寻找反应活性更好的中间体进行标记,以达到高时空分辨率。受到光动力学疗法(PDT)以及发色团诱导蛋白失活(CALI)等研究的启发,单线态氧由于其半衰期短,约为0.6微秒,且能够特异性地修饰蛋白表面氨基酸残基,作者认为单线态氧是更好的介导邻近标记的活性中间体,并发展了一种基于光激活依赖的新型邻近标记平台(PDPL)[2]。
作者将光敏蛋白和目的蛋白在细胞内进行融合表达(图1),在蓝光照射下激发产生单线态氧,从而使邻近氨基酸残基被氧化,发生极性反转,使其从亲核性氨基酸变为亲电性,进而与含有氨基的化学探针发生“接力”反应,完成邻近标记过程。化学探针上还包括一个炔基基团,可以进行后续的点击化学反应以及蛋白富集,用于LC-MS/MS表征。
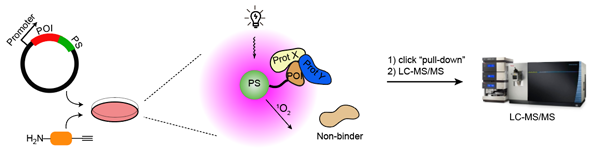
图1 PDPL流程示意图
作者认为标记特异性是此平台成功与否的关键,因此在前期进行了大量的筛选和优化。通过优化光敏蛋白以及对7种不同化学探针的优化和筛选,作者最终将标记信噪比提高到8-20倍(图2),为后续的研究奠定了基础。随后,作者通过免疫荧光成像以及各种活性氧物种的淬灭剂或稳定剂等对照实验,证明了单线态氧调控此邻近标记过程。
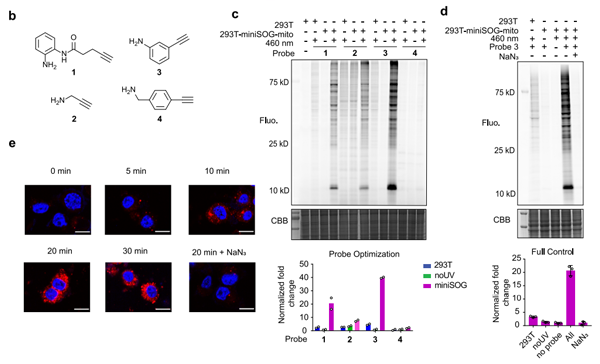
图2 PDPL标记方法的优化
为了探索标记过程的化学机理,需要确定蛋白质组中的哪些氨基酸被修饰,以及相应的修饰分子量,因此作者采用了化学蛋白质组流程TOP-ABPP方法,并利用MSFragger的FragPipe平台,进行开放式搜库(图3),发现探针特异性修饰蛋白质组的组氨酸上,偏移分子量为+229和+247(+229的水解产物),对应两次氧化产物。随后的一系列生化实验验证,包括组氨酸突变、纯化多肽的体外标记,以及人工创造的基于抗体的蛋白复合体系统的体外标记等,均可以重现上述结果。
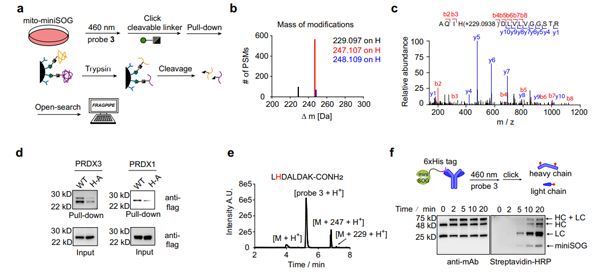
图3 PDPL标记位点的鉴定和体外标记验证
为了进一步表征PDPL方法的标记特异性,作者利用其对亚细胞器的蛋白质组进行表征(图4)。将光敏蛋白定位到不同的细胞器(线粒体,内质网,细胞核)上,凝胶电泳、免疫荧光成像以及蛋白质组学质谱均表明标记的特异性。并且PDPL方法仅需要使用正常细胞作为对照,即可完成背景扣除,而不需要像TurboID以及APEX等方法中采用的TMT标记进行多重对照实验,才能完成背景扣除。初步数据表明,与TurboID方法相比,PDPL在蛋白质组的鉴定深度以及特异性上,优于TurboID方法。
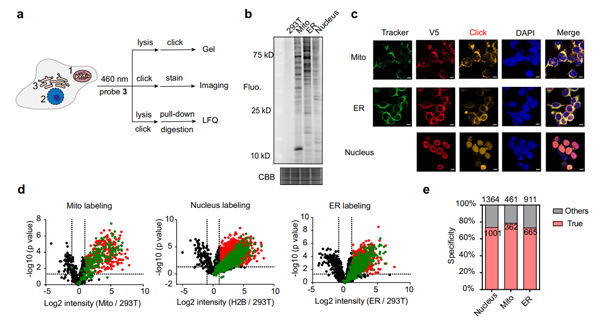
图4 PDPL在线粒体、内质网和细胞核中亚细胞器蛋白质组分析
在上述研究结果的基础上,作者尝试利用PDPL挑战细胞质蛋白的标记,因为相比于细胞膜蛋白,细胞质蛋白更加动态且包含很多瞬时相互作用,被认为是更加困难的鉴定靶点。作者首先选择了和众多疾病相关的转录共激活蛋白BRD4 (图5),源于其重要的生物学功能以及作为重要的治疗靶点的转化医学的临床意义。作者首先通过不同的光照时间来调控标记半径,展示其空间分辨率。光照条件的触发,也展示了其时间分辨的特性。GO分析结果表明,质谱中鉴定到的相互作用蛋白定位于细胞核且参与到染色质重塑以及RNA聚合酶功能。String数据库的作用网络分析表明,BRD4的互作蛋白包含了很多与HDAC相互作用的蛋白复合体,这与BRD4以及HDAC都结合乙酰化的组蛋白这一特点保持一致。除了鉴定到了众多的已知蛋白以外,作者还验证了很多未知的相互作用蛋白,Pull-down和Co-IP实验证明了PDPL方法的准确性,并鉴定到SFPQ和BRD4的相互作用,预示着BRD4介导液-液相分离的新型生物学机制。
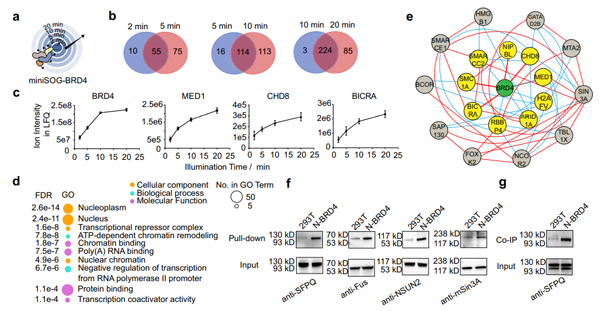
图5 PDPL分析BRD4的相互作用蛋白
最后,作者认为PDPL邻近标记方法,尤其适用于通过间接相互作用形成的、大分子量蛋白复合体中蛋白酶底物的鉴定。因此作者选择了一种与帕金森病相关的E3连接酶Parkin作为研究对象,其突变可以引起常染色体隐性青少年帕金森病。在获得蛋白质组数据的基础上,作者进行了过表达筛选,在稳定表达Parkin的细胞系中转染鉴定到的蛋白,如果转染蛋白是Parkin的作用底物,则会被降解。作者筛选到了两种新型的Parkin作用底物SNW1以及Ssu72,而其他非底物蛋白也被证明是Parkin的作用蛋白。此外,作者通过加入蛋白酶体抑制剂MG132证明这两种新型底物的降解是通过泛素化-蛋白酶体途径进行的,进一步验证了此方法的准确性。
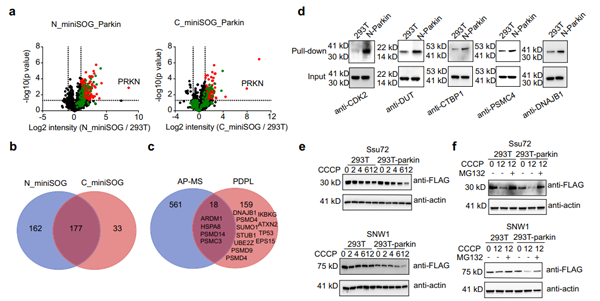
图6 PDPL结合过表达筛选鉴定Parkin底物
综上,作者发展了一种新型的基于单线态氧的邻近标记方法,该方法具有高时空分辨率,且光敏蛋白约为12 kD,约是APEX蛋白(27kD)的二分之一,约是TurboID(35kD)的三分之一,这种小尺寸蛋白可以扩展相互作用网络研究的应用范围,作者相信该方法将会具有重大的应用前景。
李刚特聘研究员为本文的唯一通讯作者,课题组研究助理翟彦生为本工作第一作者,该研究工作完全在深圳湾实验室独立完成。该研究得到了国家自然科学基金、广东省区域联合基金以及深圳湾实验室开放基金等经费支持。
李刚课题组(http://chemoproteomics-lab.szbl.ac.cn/)利用化学探针的设计及蛋白质组学技术进行化学蛋白质组学相关研究。课题组目前的研究方向包括:1)开发新型蛋白质组学方法研究蛋白-蛋白相互作用,包括邻近标记、化学交联质谱等;2)通过化学探针设计以及基于质谱的蛋白质组学研究亲核性氨基酸反应活性,从而拓展可被药物分子靶向的蛋白空间,并通过高活性氨基酸位点发现未知蛋白功能;3)研究疾病相关的蛋白质翻译后修饰,关键代谢产物、辅酶、药物分子的蛋白作用网络。目前多个研究方向均展现非常好的前景,希望早日完成相关课题的研究。
真诚邀请有志于化学蛋白质组学的科研工作者(研究助理/博士后/助理研究员)加入我们课题组,有意者请发简历到ligang@szbl.ac.cn !!!
参考文献
1. Seath, C.P., Trowbridge, A.D., Muir, T.W. &MacMillan, D.W.C. Reactive intermediates for interactome mapping. Chem. Soc. Rev.50, 2911-2926 (2021).
2. Zhai, Y., Huang, X., Zhang, K., Huang, Y., Jiang, Y., Cui, J., Zhang, Z., Chiu, C.K.C., Zhong, W.&Li, G Spatiotemporal-resolved protein networks profiling with photoactivation dependent proximity labeling. Nat. Commun. 13, 4906 (2022).
原文信息:
Spatiotemporal-resolved protein networks profiling with photoactivation dependent proximity labeling
课题组招聘:
▷ 化学蛋白质组学课题组诚聘博士后/联培博士研究生/研究助理
供稿 | 李刚课题组
编辑 | 鲍 啦
欢迎投稿、建议 | media@szbl.ac.cn