Chance favors the prepared mind
彭琴
Qin PENG
系统与物理生物学研究所
Timeline
-
2020 至今
深圳湾实验室 特聘研究员
-
2017-2020
美国加州大学圣地亚哥分校 助理项目科学家
-
2015-2017
美国加州大学圣地亚哥分校 博士后
-
2010-2015
重庆大学-美国伊利诺伊大学香槟分校(UIUC)-美国加州大学圣地亚哥分校(UCSD) 联合培养博士
-
2006-2010
重庆大学 学士
研究领域
课题组主要从事染色质活细胞成像及基因表达调控机制的研究,开发针对肿瘤等疾病相关的表观遗传修饰引起的发病机制、诊断检测和治疗的成像工具,结合深圳湾实验室医工交叉的优势,从肿瘤的表观遗传调控机理和治疗等方面共同推进研究工作。
具体研究内容:为了实时“点亮”活细胞内染色质和表观遗传修饰,课题组一直从事可遗传的新型光学成像探针的开发,来解析细胞命运转化中的力-表观调控新机制,将助力肿瘤和神经退行性疾病等重大疾病发生发展的表观遗传调控新机制研究,以及新型诊疗技术和试剂的研发。主要包括以下三方面内容:
(1)开发活细胞表观遗传修饰与染色质的新型四维(4D)标记系统;
(2)在体成像与定量细胞核与染色质上所受的机械力;
(3)开发原位功能性蛋白-DNA-RNA互作组学技术,阐明衰老、肿瘤转移等过程中的力-表观调控机制,挖掘诊疗新靶点。
成果
合成了基于全长组蛋白的荧光能量共振转移(FRET)分子探针,实现了单个活细胞水平上一种或多种组蛋白修饰的实时动态成像示踪,阐明了有丝分裂开始时H3S10ph对H3K9me3去甲基化和染色质正确重塑的调控机制。近期开发了能同时实现活细胞内单个基因位点的示踪标记和操控的生物分子探针(Simultaneous Imaging and Manipulation with Biomolecular Assemblies,SIMBA) 系统。并建立了基于定向进化技术(Directed Evolution)的高通量筛选通用平台,用于高通量构建FRET分子探针。在Nat Commun、PNAS、CELL CHEM BIOL (CELL子刊)等发表论文30余篇。
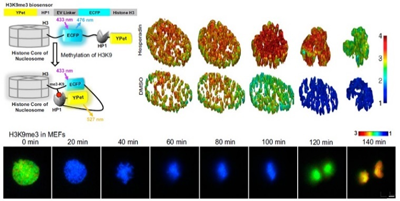
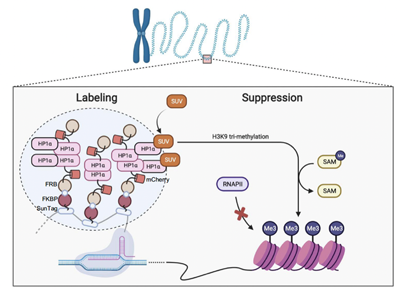
图1. 组蛋白修饰的活细胞成像 图2. 单基因位点的示踪标记和操控
荣誉
• 2020 深圳市孔雀计划人才
• 2022 广东省珠江人才计划青年拔尖人才项目
媒体报道
代表论文
[1] Peng Q. *, †, Huang Z. *, Sun K., Liu Y., Yoon C., Harrison R., Schmitt D., Zhu L., Wu Y., Tasan I., Zhao H., Zhang J., Zhong S., Wang Y†. Engineering Inducible Biomolecular Assemblies for Genome Imaging and Manipulation in Living Cells. Nat Commun, 2022, 13(1): 7933. DOI: 10.1038/s41467-022-35504-x. († Corresponding author, Featured Article)
[2] Harrison R., Weng K., Wang Y.†, Peng Q.†. Phase Separation and Histone Epigenetics in Genome Regulation. Curr Opin Solid St M. 2021, 25(1):100892. doi: 10.1016/j.cossms.2020.100892. († Corresponding author)
[3] Huang Z, Ouyang M., Lu S., Wang Y†, Peng Q.†. Optogenetic Control for Investigating Subcellular Localization of Fyn Kinase Activity in Single Live Cells. J Mol Biol. 2020, pii: S0022-2836(20)30236-9. DOI: 10.1016/j.jmb.2020.03.015. († Corresponding author)
[4] Peng Q., Lu S., Shi Y., Pan Y., Limsakul P., Chernov AV., Qiu J., Chai X., Shi Y., Wang P., Ji Y., Li YJ., Strongin AY., Verkhusha VV., Izpisua Belmonte JC., Ren B., Wang Y., Chien S. †, Wang Y†. Coordinated Histone Modifications and Chromatin Reorganization in a Single Cell Revealed by FRET Biosensors. Proc Natl Acad Sci U S A. 2018, 115(50): E11681-E11690. doi: 10.1073/pnas.
[5] Limsakul P. *, Peng Q. *, Wu Y., Allen ME., Liang J., Remacle AG., Lopez T., Ge X., Kay BK., Zhao H., Strongin AY., Yang XL., Lu S., Wang Y †. Directed Evolution to Engineer Monobody for FRET Biosensor Assembly at Live-Cell Surface. Cell Chem Biol. 2018, 25(4): 370-379.e4. doi: 10.1016/j.chembiol.2018.01.002. * Equal contribution.