
近日,深圳湾实验室系统与物理生物学研究所李磊团队在Genome Medicine上发表了题为Multimodal-based analysis of single-cell ATAC-seq data enables highly accurate delineation of clinically relevant tumor cell subpopulations的研究论文。该研究开发了一种基于单细胞表观组学的多模态整合算法MAAS,实现了对临床相关肿瘤细胞亚群的高精度识别。
在肿瘤进展过程中,癌细胞不断积累遗传与表观遗传改变,逐步分化为具有不同分子特征的细胞亚群,从而形成高度复杂的瘤内异质性[1]。单细胞RNA测序(scRNA-seq)虽然能够在一定程度上区分恶性与非恶性细胞,但由于其主要反映转录活性,往往难以捕捉那些不直接体现在基因表达变化中、却对临床结局具有关键影响的细胞亚群[2]。相比之下,单细胞表观基因组学技术(如 scATAC-seq)能够揭示染色质可及性等核心调控信息,为深入解析肿瘤异质性提供了一个新的高分辨率视角。遗传与表观遗传变化的协同作用是推动肿瘤演化和治疗耐药的核心动力,这凸显了对多维分子信息进行系统整合的必要性[3]。然而,现有方法未充分挖掘 scATAC-seq 数据中同时蕴含的遗传与表观遗传信号,也难以解析拷贝数异质性较低但生物学意义重大的细胞亚群;再加上 scATAC-seq 数据本身高度稀疏且噪声显著,这些问题共同构成了当前解析肿瘤异质性的关键瓶颈,并限制了其生物学解释及在临床转化中的潜力。
近日,深圳湾实验室系统与物理生物学研究所李磊团队在Genome Medicine上发表了题为Multimodal-based analysis of single-cell ATAC-seq data enables highly accurate delineation of clinically relevant tumor cell subpopulations的研究论文。该研究开发了一种基于单细胞表观组学的多模态整合算法MAAS,实现了对临床相关肿瘤细胞亚群的高精度识别。

MAAS提出了一种全新的凸优化数学模型,旨在系统整合染色质开放性、体细胞点突变和拷贝数变异等多维度信息。其核心流程为:先校正拷贝数变异对染色质开放性定量的影响;继而基于各种数据类型构建细胞相似性矩阵;进而设计包含自表达项的非负矩阵分解损失函数,并通过块坐标下降法求解最优子空间;最终结合自适应K-means法对子空间进行聚类,从而识别细胞亚群并推断其层次结构。此外,算法还可通过系数矩阵的秩,定量评估不同特征在细胞亚群区分中的相对贡献(图1)。
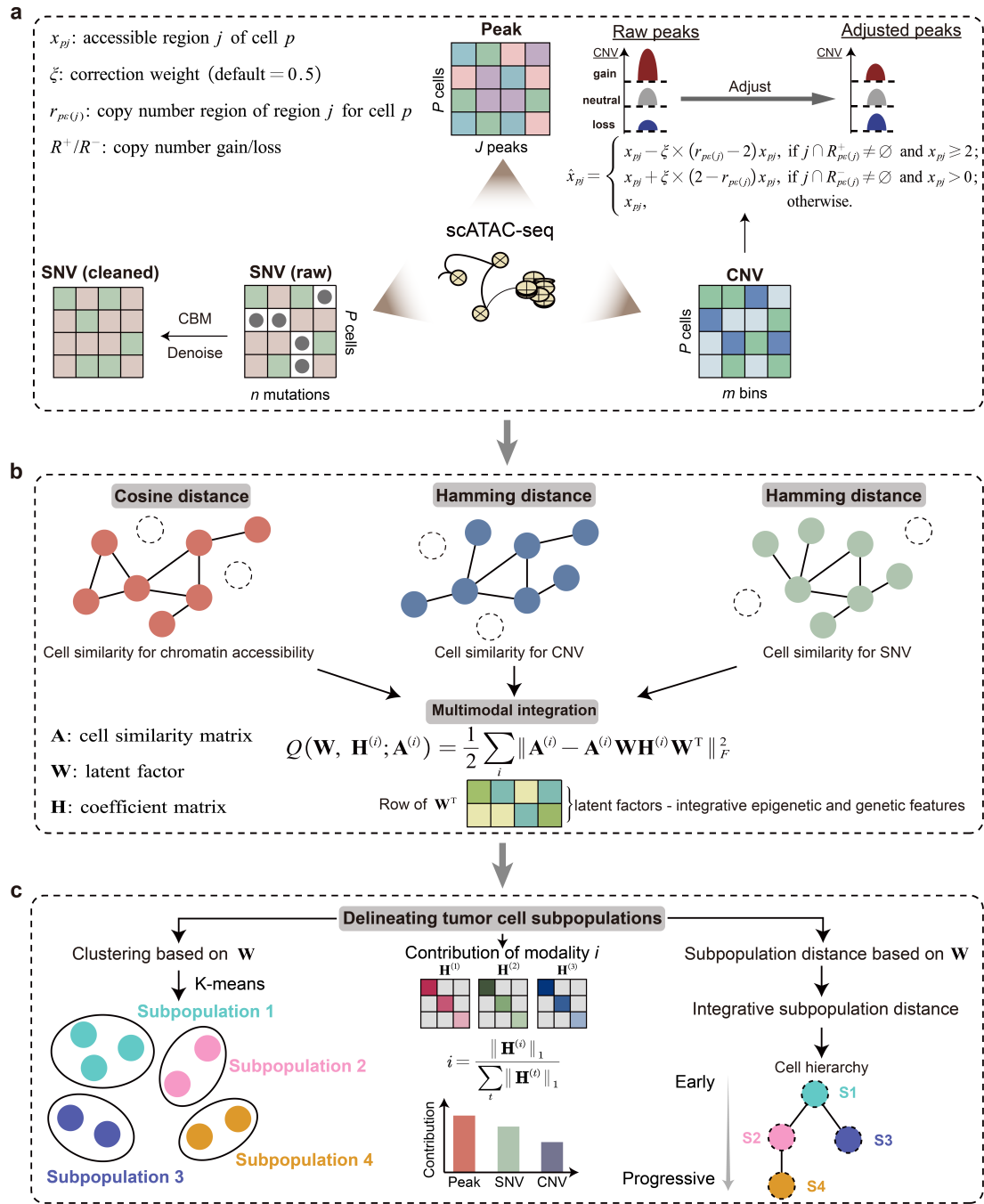
图1 MAAS计算框架
基准测试结果表明,在多个模拟与真实数据集上,MAAS在细胞亚群识别准确性方面显著优于单模态分析方法及现有多组学整合工具。研究团队进一步将MAAS应用于拷贝数变异水平较低的儿童颅后窝室管膜瘤和B细胞淋巴瘤数据,成功识别出传统方法难以检测的耐药细胞亚群。而在高度恶性的胶质瘤中,MAAS识别出一个对替莫唑胺治疗表现出显著耐药性的新型细胞亚群C2,并通过体外敲低、过表达及rescue实验,进一步验证了其耐药特性(图2)。此外,研究团队基于MAAS构建的预后分子签名,在泛癌层面上能更准确地预测患者生存结局,其预测性能显著优于传统临床病理指标及已有分子签名。
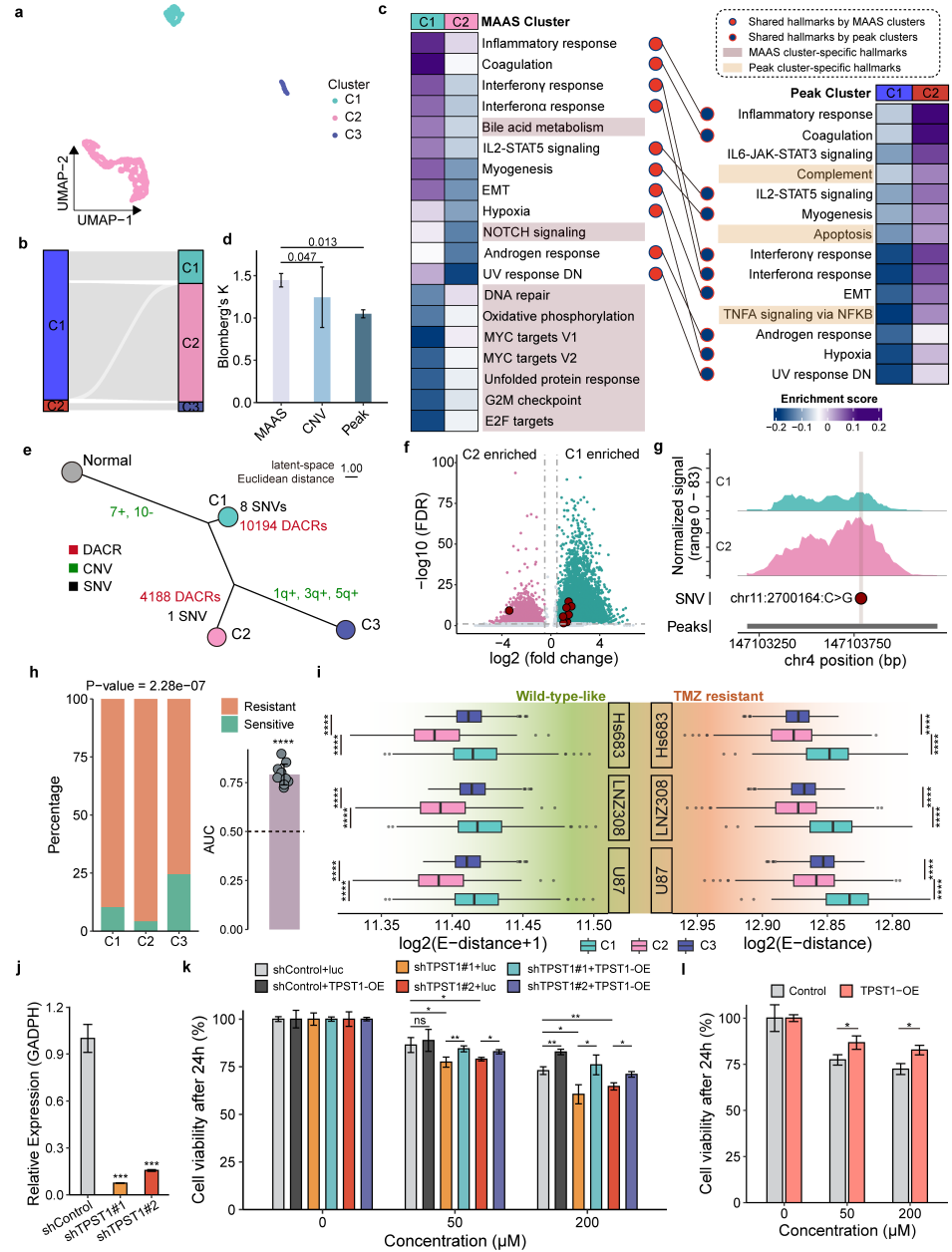
图2 MAAS识别新型胶质瘤细胞亚群及其对替莫唑胺的耐药性
综上所述,该研究为单细胞表观组学在肿瘤异质性解析中的应用提供了全新的计算框架,未来有望推广至更广泛的单细胞多组学数据与其他疾病研究领域,同时也为细胞治疗策略的开发提供潜在靶点。
本论文共同第一作者为李磊课题组博士生熊可为与于晨课题组助理研究员汪伟博士;李磊研究员为最后通讯作者,于晨研究员为共同通讯作者;研究团队成员丁若凡博士、罗丁琳、覃杨梅博士、邹旭东博士等亦对本研究作出重要贡献。本研究得到了香港科技大学王吉光教授的大力支持,相关计算分析工作于深圳湾实验室高性能计算平台完成。
李磊课题组长期致力于通过开发和运用新的计算生物学方法,整合多种大规模临床数据解析人类复杂疾病的调控机理与转化应用。课题组长期招聘博士后和科研助理等岗位,详情请见课题组网站https://bioinfo.szbl.ac.cn/index.html。
参考文献
1. Mazor, T. et al. DNA Methylation and Somatic Mutations Converge on the Cell Cycle and Define Similar Evolutionary Histories in Brain Tumors.Cancer Cell28, 307–317 (2015).
2. Corces, M.R. et al. Lineage-specific and single-cell chromatin accessibility charts human hematopoiesis and leukemia evolution.Nat Genet48, 1193–1203 (2016).
3. Nam, A.S., Chaligne, R. & Landau, D.A. Integrating genetic and non-genetic determinants of cancer evolution by single-cell multi-omics.Nat Rev Genet22, 3–18 (2021).
原文信息:
Multimodal-based analysis of single-cell ATAC-seq data enables highly accurate delineation of clinically relevant tumor cell subpopulations
文章来源|李磊课题组
编辑|鲍 啦
责编|远 山
欢迎投稿、建议|media@szbl.ac.cn

 粤公网安备44031102000926号
粤公网安备44031102000926号