
2022年7月23日,深圳湾实验室李磊课题组在Cell子刊STAR Protocols 杂志在线发表了题为“Using population-scale transcriptomic and genomic data to map 3′UTR alternative polyadenylation quantitative trait loci”的分析方案文章,开发了一个用户友好的,可广泛应用的基于大规模的标准转录组测序数据和对应的基因组变异信息识别3′aQTL的分析工具(3aQTL-pipe)。
全基因组关联分析(GWAS)已经发现成千上万与人类性状和常见疾病相关的功能性非编码区遗传变异(SNP)位点[1]。然而,这些 SNP 的功能解释仍然是一个重大的挑战。选择性多聚腺苷酸化(APA)作为一种重要的RNA转录后调控机制,广泛存在于所有真核生物中。APA异常调控会影响目标mRNA的稳定性和翻译效率以及蛋白质的细胞定位,进一步影响发育和疾病(如癌症)的发生发展[2,3]。课题组前期提出了位于基因的3′UTR的选择性多聚腺苷酸化(APA)关联的遗传变异位点(3′aQTL)可以解释约16.1%的人类复杂疾病和性状差异,为揭示人类复杂性状和疾病病因学提供了新的途径。但是,基于大规模转录组数据和基因组数据分析APA水平及识别APA水平差异相关的遗传变异位点的流程十分复杂且依赖大量的专业知识和经验,不利于该方法的广泛应用。
2022年7月23日,深圳湾实验室李磊课题组在Cell 子刊STAR Protocols 杂志在线发表了题为“Using population-scale transcriptomic and genomic data to map 3′UTR alternative polyadenylation quantitative trait loci”的分析方案文章,开发了一个用户友好的,可广泛应用的基于大规模的标准转录组测序数据和对应的基因组变异信息识别3′aQTL的分析工具(3aQTL-pipe)。该工具可通过简单的命令即可并行处理大规模转录组数据并分析APA的定量水平,并且可通过简单的命令进行下游的APA关联的遗传变异位点分析。该工具不依赖于特定物种的参考信息,可被广泛应用于其他数据集或其他物种的数据。
在这篇文章中,作者以GEUVADIS RNA-seq项目中的欧洲人群样本为示例系统地描述了使用3aQTL-pipe的分析流程及命令行参数,包括软件及硬件环境配置,所需数据集的获取,分析结果的解析,详尽的代码和文件操作说明,以及分析流程中可能遇到的问题及解决方案。3aQTL-pipe基于python3和R语言进行开发的,包含三个主要的模块,分别是:1)大规模样本的APA定量分析;2)识别APA水平变化相关联的遗传变异位点(3‘aQTL);3)对3’aQTL进行精细分析以鉴定出可能的因果变异位点。为了方便研究者使用,作者已经将3aQTL-pipe的代码部署到Github上(https://github.com/3UTR/3aQTL-pipe)和zenodo网站上(https://zenodo.org/record/6658555#.YtrRNuxBzLA)。
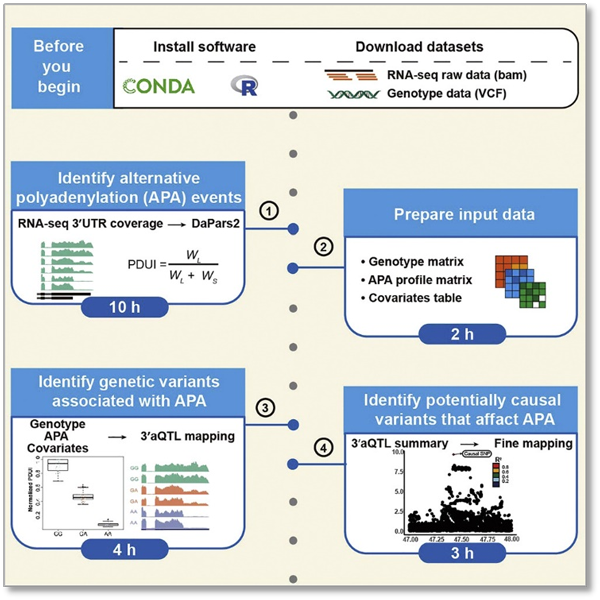
图1 识别3'非翻译区选择性多聚腺苷酸化数量性状位点的分析方案
深圳湾实验室系统物理与生物学研究所李磊课题组助理研究员邹旭东和博士后丁若凡为该工作的共同第一作者,深圳湾实验室系统物理与生物学研究所李磊研究员为该工作的通讯作者。加州大学尔湾分校李蔚教授为共同通讯作者。该研究得到了国家自然科学基金、NIH National Institute on Aging grant和Thompson Family Foundation (TAME-AD)项目支持,以及深圳湾实验室高性能计算与信息平台的支持,同时也感谢哥伦比亚大学王告教授的贡献。
参考文献
[1] Loos, R. J. F. 15 years of genome-wide association studies and no signs of slowing down. Nat Commun 11, 5900, doi:10.1038/s41467-020-19653-5 (2020).
[2] Mitschka, S. & Mayr, C. Context-specific regulation and function of mRNA alternative polyadenylation. Nat Rev Mol Cell Biol, doi:10.1038/s41580-022-00507-5 (2022).
[3] Tian, B. & Manley, J. L. Alternative polyadenylation of mRNA precursors. Nat Rev Mol Cell Biol 18, 18-30, doi:10.1038/nrm.2016.116 (2017).
原文信息:
Using population-scale transcriptomic and genomic data to map 3′ UTR alternative polyadenylation quantitative trait loci
文章来源 | 岚翰生命科学
编辑 | 鲍 啦
欢迎投稿、建议 | media@szbl.ac.cn

 粤公网安备44031102000926号
粤公网安备44031102000926号