
2024年6月18日,深圳湾实验室梁卓斌和耶鲁大学Farren J. Isaacs团队合作在Nature Communications杂志在线发表了题为Enhanced eMAGE applied to identify genetic factors of nuclear hormone receptor dysfunction via combinatorial gene editing的研究论文。该研究对eMAGE技术进行了迭代优化,显著提高了编辑效率和范围,并开发了可调控的错配修复抑制系统,将增强型eMAGE技术成功应用于研究人类核激素受体功能障碍。这项工作为研究具有复杂遗传基础或需要高效多重编辑的生物系统特性(包括合成基因组工程)提供了一种有价值的基因组编辑方法。
基因组编辑技术在研究基因型和表型的因果关系、重新编程细胞行为以及设计合成基因组等方面发挥了重要作用【1,2】。然而,现有的基因组编辑技术在实现大规模、高精度以及多重编辑方面仍存在不足,例如脱靶效应、细胞毒性、编辑精度和多重编辑上限等【3】。传统的基因组编辑技术使用可编程核酸酶在基因组中产生位点特异性DNA双链断裂(DSB),然后通过内源性机制修复DSB,从而实现基因编辑。然而,这些方法在需要生成多位点(>10)、具有碱基对水平精度的组合基因组修饰时存在不足【4】。
为了克服这些限制,2017年,Farren J. Isaacs团队首次报道了真核生物多重基因组工程(eMAGE)技术【5】。eMAGE是一种不依赖DSB的基因组编辑技术,它利用合成单链寡脱氧核苷酸(ssODN)靶向目标序列DNA复制叉的滞后链,以及规避DNA错配修复(MMR)来实现多重基因组编辑。与传统的基于DSB的基因编辑工具相比,eMAGE具有更高的编辑效率和更低的细胞毒性,并且可以同时编辑多个基因组位点【5】。然而,初始版本的eMAGE技术在编辑效率、编辑距离和基因组稳定性等方面仍有改进空间。
2024年6月18日,深圳湾实验室梁卓斌和耶鲁大学Farren J. Isaacs团队合作在Nature Communications杂志在线发表了题为Enhanced eMAGE applied to identify genetic factors of nuclear hormone receptor dysfunction via combinatorial gene editing的研究论文。该研究对eMAGE技术进行了迭代优化,显著提高了编辑效率和范围,并开发了可调控的错配修复抑制系统,将增强型eMAGE技术成功应用于研究人类核激素受体功能障碍。这项工作为研究具有复杂遗传基础或需要高效多重编辑的生物系统特性(包括合成基因组工程)提供了一种有价值的基因组编辑方法。
为了实现快速和组合的多重编辑,研究人员首先重新设计了eMAGE报告基因,采用URA3(FS)-RFP(FS)双荧光报告基因系统,并通过流式细胞术分析RFP阳性细胞的频率来更准确、快速地评估eMAGE等位基因替换频率(ARF),从而显著缩短了筛选周期,提高了检测通量,加速了eMAGE工作流程。此外,研究人员还系统地探究了单链寡脱氧核苷酸(ssODN)对eMAGE编辑效率的影响,发现通过优化ssODN浓度、靶向编辑位点与共选择标记的ssODN比例,以及对ssODN末端核苷酸进行硫代磷酸酯键修饰等策略,可以显著提高eMAGE的编辑效率,最高可达~48%。
研究人员假设在目标基因组区域两侧引入两个共选择标记可以维持较长基因组位点上的高水平多位点编辑。为了验证这一假设,他们构建了一个包含三个荧光报告基因和两个带有预定义移码突变的选择标记的五基因结构。结果表明,使用双重共选择策略后,三个荧光报告基因的eMAGE ARF均显著提高(50-70%),并且显著富集了双重和三重编辑的细胞,减少了未编辑的亚群。此外,研究人员还发现,双重标记共选择可以将eMAGE编辑距离扩展到20 kb以上,同时保持较高的编辑水平。
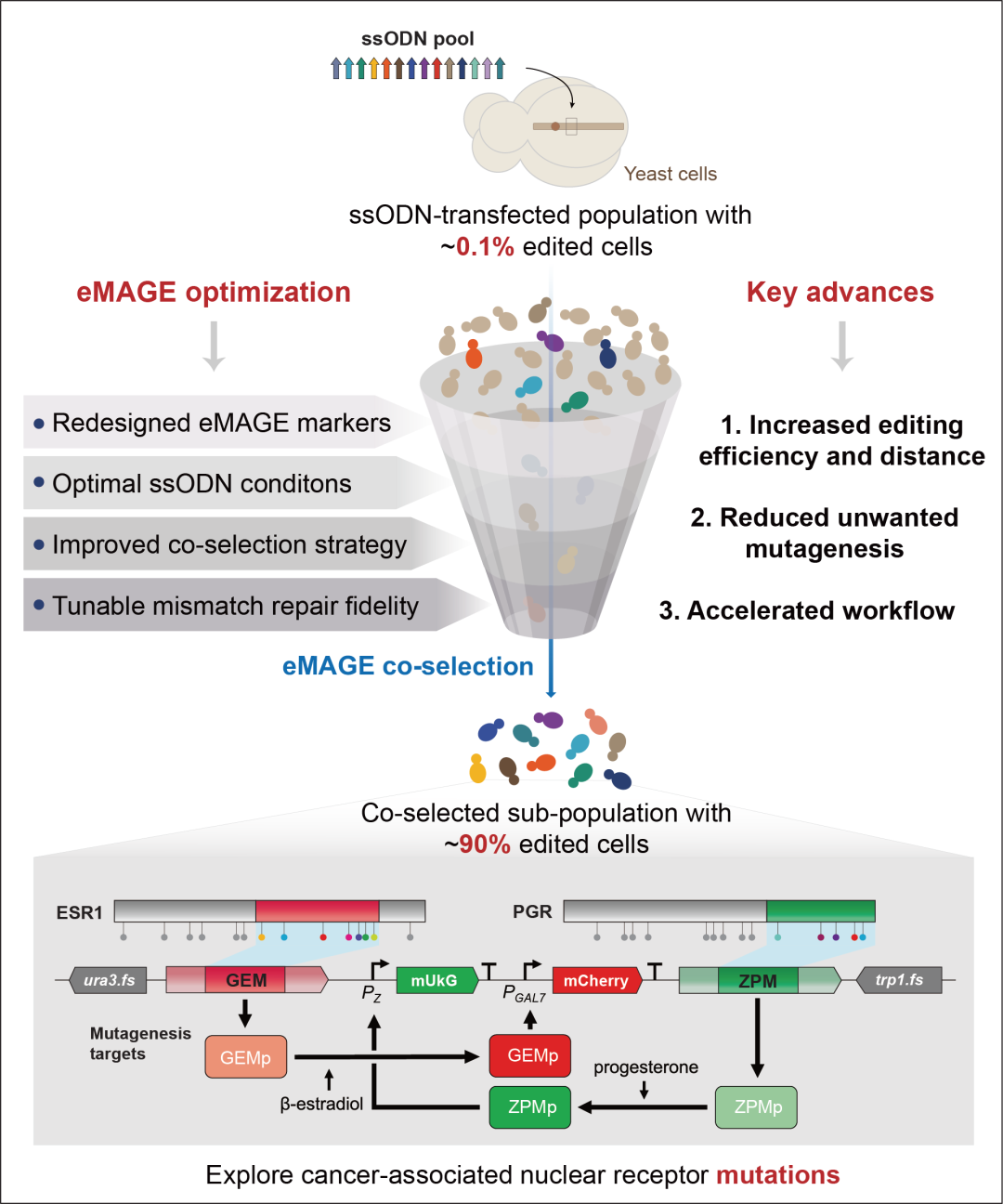
为了在保持高eMAGE ARF的同时避免永久性MMR失活带来的基因组不稳定风险,研究人员开发了一种在DNA修复能力正常的细胞中瞬时可控的错配修复抑制系统。通过筛选和优化MMR-DN突变体,他们发现特定的MSH2突变体在较高表达水平下或与MLH1-WT在较低表达水平下组合表达时,能实现与msh2Δ细胞相似的移码校正频率和多重编辑水平(高达~90%)。此外,Luria-Delbrück波动分析显示,诱导型错配修复抑制系统的自发突变率比错配修复敲除低17倍,接近野生型细胞的水平。该系统提高了被编辑细胞的基因组稳定性,降低了DNA复制过程中背景突变的产生。
在应用方面,研究人员利用增强型eMAGE技术在酿酒酵母中构建了人类雌激素受体α (ERα) 和孕酮受体 (PR) 配体结合域 (LBDs) 中的癌症相关突变库。通过对回收的细胞群进行NGS测序和富集分析,研究人员观察到GEM和ZPM变体的富集,并通过克隆表征和转录活性分析,鉴定出一些在临床上尚未观察到的具有组成型活性的变体。这些结果表明,eMAGE技术可以用于研究核受体功能障碍,并识别导致受体组成型激活的突变。
本研究对eMAGE基因组编辑技术进行系统升级,显著提高了编辑效率和精确度,并成功应用于研究人类核激素受体功能障碍,为基因组编辑技术在生物医学研究和临床应用方面提供了新的思路和工具。
深圳湾实验室梁卓斌特聘研究员和耶鲁大学Farren J. Isaacs教授为该论文的共同通讯作者,梁卓斌研究员和耶鲁大学博士生Peter N. Ciaccia为论文的共同第一作者。
参考文献
1. Chari, R. & Church, G. M. Beyond editing to writing large genomes. Nat Rev Genet 18, 749-760, doi:10.1038/nrg.2017.59 (2017).
2. Anzalone, A. V., Koblan, L. W. & Liu, D. R. Genome editing with CRISPR–Cas nucleases, base editors, transposases and prime editors. Nature biotechnology 38, 824-844, doi.org/10.1038/s41587-020-0561-9 (2020).
3. Thompson, D. B. et al. The Future of Multiplexed Eukaryotic Genome Engineering. ACS Chem Biol 13, 313-325, doi:10.1021/acschembio.7b00842 (2018).
4. McCarty, N. S., Graham, A. E., Studena, L. & Ledesma-Amaro, R. Multiplexed CRISPR technologies for gene editing and transcriptional regulation. Nature communications 11, 1281, doi:10.1038/s41467-020-15053-x (2020).
5. Barbieri, E. M., Muir, P., Akhuetie-Oni, B. O., Yellman, C. M. & Isaacs, F. J. Precise Editing at DNA Replication Forks Enables Multiplex Genome Engineering in Eukaryotes. Cell 171, 1453-1467 e1413, doi:10.1016/j.cell.2017.10.034 (2017).
原文信息:
Enhanced eMAGE applied to identify genetic factors of nuclear hormone receptor dysfunction via combinatorial gene editing
欢迎投稿、建议 | media@szbl.ac.cn

 粤公网安备44031102000926号
粤公网安备44031102000926号