
2024年7月10日,深圳湾实验室系统与物理生物学研究所李磊与复旦大学倪挺团队在Science Advances杂志发表题为“Pan-cancer transcriptome analysis reveals widespread regulation through alternative tandem transcription initiation”的研究论文。该研究开发了首个可以从转录组识别基因可变串联转录起始的新方法,并利用该方法系统解析了人类癌症中可变串联转录起始介导信使RNA的5′ UTR长度变化规律,并探究了其介导的基因表达调控在癌症发生发展过程中的作用,为理解癌症发生机制提供新角度。
基因组变异和表观遗传层面的改变大大增加了癌细胞转录组的复杂性。基因的可变转录起始主要分为两种:可变第一个外显子和可变串联转录起始。可变第一个外显子指从相距较远的不同启动子转录起始产生不同第一个外显子的转录本,而可变串联转录起始指从复合型转录起始区域的不同串联转录起始位点转录起始产生5′非翻译区(5′ UTR)长短不同的转录本(图1A)。已有多项研究报道可变第一个外显子介导的基因表达调控参与癌症发生发展(1-3)。然而,在癌症发生发展过程中可变串联转录起始位点使用的动态变化及其介导的基因表达调控的生物学作用尚不清楚。
2024年7月10日,深圳湾实验室李磊团队与复旦大学生命科学学院倪挺团队联合在Science Advances在线发表了题为 Pan-cancer transcriptome analysis reveals widespread regulation through alternative tandem transcription initiation的研究论文,系统解析了人类癌症中可变串联转录起始介导信使RNA的5′ UTR长度变化规律,并探究了其介导的基因表达调控在癌症发生发展过程中的作用,为理解癌症发生机制提供新角度。
作者首先从FANTOM5项目、GENCODE数据库和50对肝癌及其配对正常组织样本CAGE数据中共收集到超过30万个转录起始位点(TSS),进一步比较分析发现约90%的蛋白质编码基因拥有多个串联转录起始位点,该比例远高于拥有可变第一个外显子的蛋白质编码基因比例(33.5%),说明蛋白质编码基因的表达受到可变串联转录起始的广泛调控。
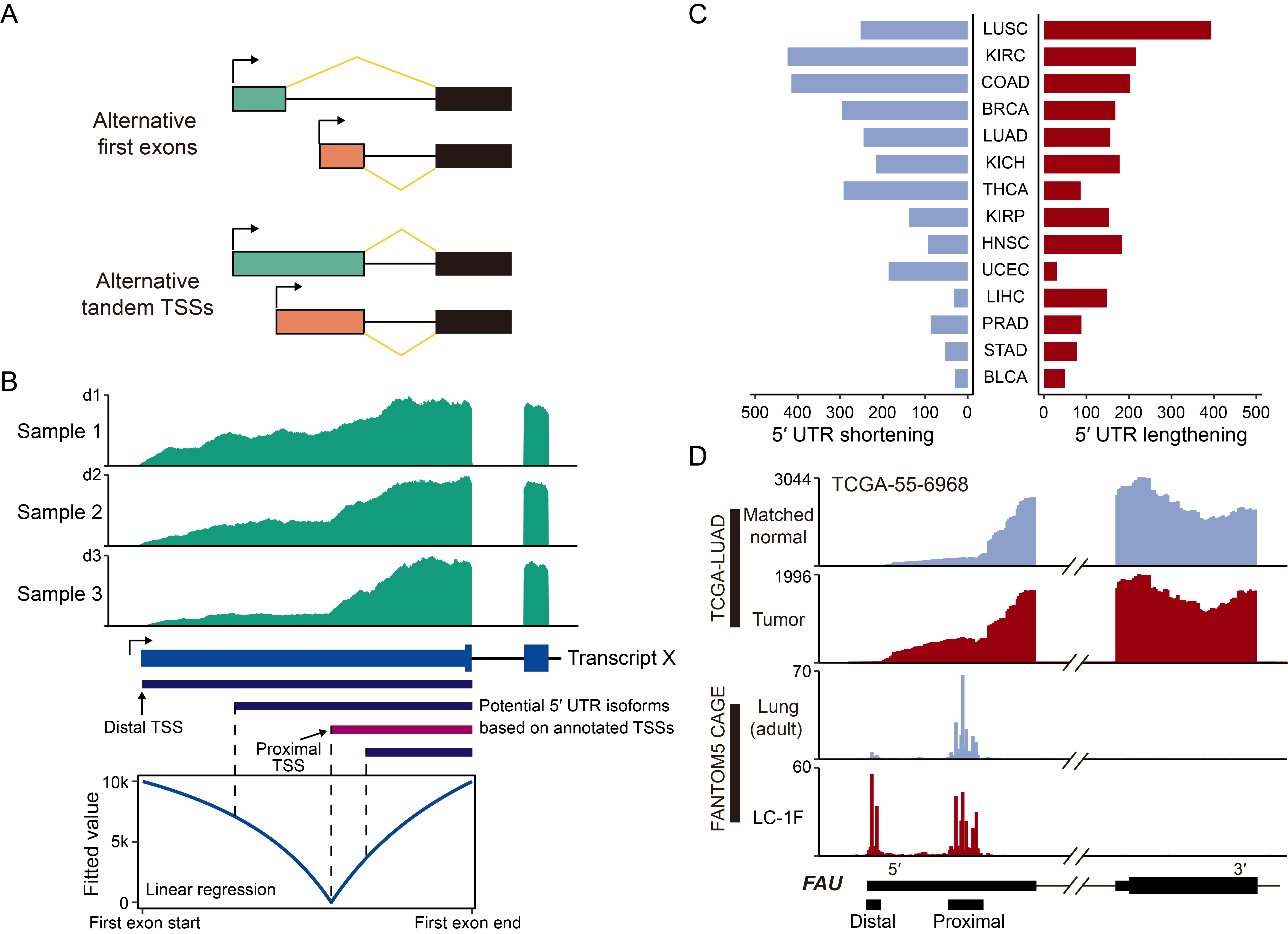
图1 利用常规RNA-seq数据解析可变串联转录起始的动态调控
为了利用海量公开发表的常规RNA-seq数据解析可变串联转录起始事件的规律特征,作者开发了算法DATTSS,并从多个层面评估了DATTSS的可靠性。算法DATTSS以收集到的转录起始位点为先验信息,利用线性回归模型对转录本第一个外显子区域的RNA-seq读段覆盖度进行建模,通过识别最佳分割点来推断串联转录起始位点进而定量其使用(图1B)。作者将DATTSS算法应用在TCGA数据库收录的14种癌症类型的RNA-seq数据上,分别鉴定到80~646个基因在癌/癌旁样本中发生显著失调的可变串联转录起始(图1C)。以基因FAU为例,相比于癌旁样本,其在肺癌样本中更多地使用远端TSS,呈现5′ UTR变长趋势(图1D)。有趣的是,作者发现不同癌症类型中66.6~85.6%的受到差异可变串联转录起始影响的基因不受到其基因表达或差异可变第一个外显子层面的影响,说明可变串联转录起始是癌症发生过程中基因表达的新调控因素。通过整合多组学数据,作者发现可变串联转录起始受到表观遗传层面(如DNA甲基化)的调控,表观遗传抑制剂(DAC或SB939)处理会在全局层面诱导可变串联转录起始的动态变化。作者挑选多个基因进行分子实验,发现可变串联转录起始所产生的5′ UTR长短不同的转录本,具有不同的稳定性或翻译效率,可能对最终的蛋白产量存在潜在影响。
作者以基因TIMM13(编码线粒体内膜转位酶)为例,阐明了其5′ UTR变短介导的蛋白表达上调在肺癌中的作用。在肺癌样本中TIMM13相比于配对癌旁样本近端TSS使用比例显著增加,呈现5′ UTR变短趋势。结合Ribosome profiling数据和Luciferase实验,作者证实5′ UTR较短TIMM13转录本能逃脱uORF的抑制效应,产生更多的蛋白质。在肺癌细胞NCI-H1299和A549中敲低TIMM13会损伤线粒体功能,抑制肺癌细胞的增殖和迁移能力。作者针对TIMM13的两个TSS设计不同的sgRNA,利用CRISPRa激活其转录,发现靶向TIMM13近端TSS的sgRNA能特异激活近端TSS的转录(一定程度上模拟了肺癌中近端TSS使用比例增加的现象),且该sgRNA能上调TIMM13蛋白水平,并促进肺癌细胞的增殖和迁移能力。
深圳湾实验室邹旭东博士与复旦大学人类表型组研究院博士后赵昭昭、博士生陈宇、林丽敏为本文的共同第一作者,深圳湾实验室李磊研究员与复旦大学生命科学学院/人类表型组研究院倪挺教授为共同通讯作者。
参考文献
1. D. Demircioğlu et al., A Pan-cancer Transcriptome Analysis Reveals Pervasive Regulation through Alternative Promoters. Cell 178, 1465-1477.e1417 (2019).
2. N. M. Shah et al., Pan-cancer analysis identifies tumor-specific antigens derived from transposable elements. Nature genetics, (2023).
3. M. Zhang et al., Integrative analysis of ultra-deep RNA-seq reveals alternative promoter usage as a mechanism of activating oncogenic programmes during prostate cancer progression. Nat Cell Biol, (2024).
原文信息:
Pan-cancer transcriptome analysis reveals widespread regulation through alternative tandem transcription initiation
欢迎投稿、建议 | media@szbl.ac.cn

 粤公网安备44031102000926号
粤公网安备44031102000926号