
电话:+86-755-86967710
邮箱:webmaster@szbl.ac.cn

肿瘤研究所
leah.zhanglei@szbl.ac.cn
癌症生物学,免疫学,计算生物学
特聘研究员
Tenure-track 助理教授
博士后
博士
学士
张雷课题组致力于整合患者和小鼠模型的单细胞多组学分析及体内外功能研究,以从分子和细胞层面解析肿瘤以及免疫相关疾病的发病机理,探究免疫疗法的响应及抵抗机制,探索肿瘤发生/治疗响应相关细胞的功能及下一代靶向策略。
张雷,深圳湾实验室特聘研究员,北京大学深圳研究生院博士生导师。国家自然科学基金优秀青年基金获得者,国家重点研发计划青年科学家项目首席。博士毕业于北京大学分子医学研究所,2016年加入北京大学生物医学前沿创新中心(BIOPIC)进行博士后训练。近年来主要从事消化道疾病微环境单细胞功能组学及治疗策略开发应用研究,包括系统性地解析结直肠癌浸润T细胞和髓系细胞的特征及动态关系,揭示不同靶向髓系细胞免疫治疗策略的作用机理,解析炎症性肠病单细胞水平特征和潜在治疗靶点。研究成果以第一作者发表在Nature和Cell,以通讯作者发表在Nature Computational Science上。相关成果获评中国生物信息学十大进展,Cell年度最佳学术论文,北京市科学技术奖自然科学奖一等奖(共同完成人)等。
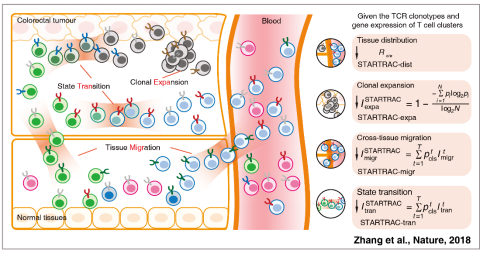
张雷博士与团队利用TCR受体(TCR)作为标签,整合单细胞转录组信息,开发了STARTRAC生物信息学方法,对结直肠癌患者癌组织、癌旁组织以及外周血中鉴定的20类不同类型T细胞的组织分布、克隆增生、组织迁移和状态变化特征进行定量刻画。此工作突破人体内T细胞动态变化关系难以定量追踪的难点,相关成果在Nature杂志发表,并获得2018年中国生物信息学十大进展:
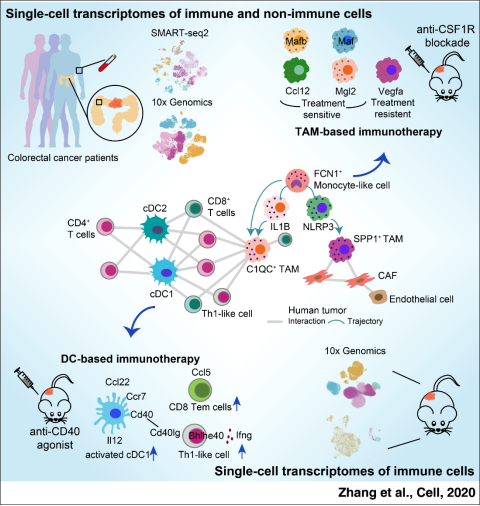
张雷博士与团队首次在单细胞水平对结直肠癌浸润髓系细胞类群进行了系统性的刻画,解析了肿瘤相关巨噬细胞的类群特征、谱系发育及细胞间相互作用关系,同时利用小鼠模型,对anti-CSF1R抑制剂和anti-CD40激动剂两种靶向髓系细胞的免疫治疗策略潜在的作用机理给出了解释。此工作建立了整合肿瘤患者和小鼠肿瘤模型,在单细胞水平研究肿瘤免疫治疗机理及应用的范例。相关成果被Cell杂志评选为2020年最佳医学研究论文,是中国地区2项成果之一:
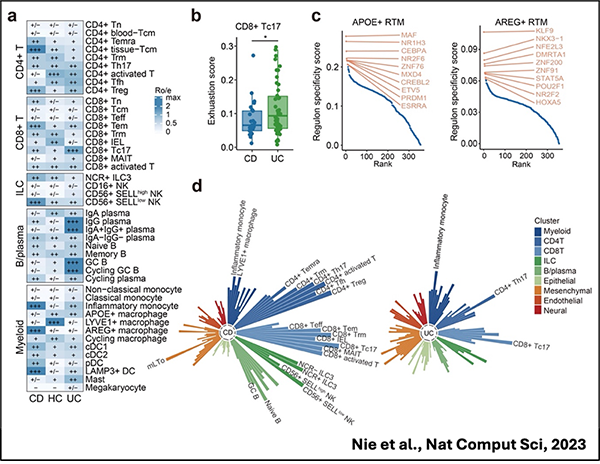
张雷团队系统性地揭示了炎症性肠病(IBD)单细胞水平特征和潜在治疗靶点。该研究对来自IBD患者的400多个样本单细胞转录组数据集进行了系统的整合分析,并结合GWAS研究及药物临床试验信息,揭示了IBD的细胞亚群和潜在的治疗靶点,开发了交互式在线分析平台scIBD(http://scibd.cn/),为IBD研究领域提供了宝贵的资源。同期Nature Computational Science杂志刊发了相关评述文章:
1.Lei Zhang#; Xin Yu#; Liangtao Zheng#; Yuanyuan Zhang#; Yansen Li; Qiao Fang; Ranran Gao; Boxi Kang; Qiming Zhang; Julie Y. Huang; Hiroyasu Konno; Xinyi Guo; Yingjiang Ye; Songyuan Gao; Shan Wang; Xueda Hu; Xianwen Ren; Zhanlong Shen*; Wenjun Ouyang*; Zemin Zhang*;Lineage tracking reveals dynamic relationships of T cells in colorectal cancer.Nature, 2018, 564(7735): 268-272.
2.Lei Zhang; Zemin Zhang*;Recharacterizing tumor-infiltrating lymphocytes by single-cell RNA sequencing.Cancer Immunology Research, 2019, 7(7): 1040-1046.
3.Lei Zhang#; Ziyi Li#; Katarzyna M. Skrzypczynska#; Qiao Fang; Wei Zhang; Sarah A. O'Brien; Yao He; Lynn Wang; Qiming Zhang; Aeryon Kim; Ranran Gao; Jessica Orf; Tao Wang; Deepali Sawant; Jiajinlong Kang; Dev Bhatt; Daniel Lu; Chi-Ming Li; Aaron S. Rapaport; Kristy Perez; Yingjiang Ye; Shan Wang; Xueda Hu; Xianwen Ren; Wenjun Ouyang; Zhanlong Shen*; Jackson G. Egen*; Zemin Zhang*; Xin Yu*;Single-cell analyses inform mechanisms of myeloid-targeted therapies in colon cancer.Cell,2020, 181(2): 442-459.
4. Xianwen Ren#;Lei Zhang#; Yuanyuan Zhang; Ziyi Li; Nathan Siemers; Zemin Zhang*;Insights gained from single-cell analysis of immune cells in the tumor microenvironment.Annual Review of Immunology,2021, 39: 583-609.
5. Hu Nie; Peilu Lin; Yu Zhang; Yihong Wan; Jiesheng Li; Chengqian Yin;Lei Zhang*;Single-cell meta-analysis of inflammatory bowel disease with scIBD.Nature Computational Science, 2023, 3: 522–531.
联系方式
地址:深圳市光明区光侨路高科创新中心
电话:+86-755-86967710
邮箱:webmaster@szbl.ac.cn
邮编:518132
Copyright © 2024 深圳湾实验室 版权所有www.szbl.ac.cn All Rights Reserved.粤ICP备19032884号 粤公网安备44031102000926号
粤公网安备44031102000926号


