
电话:+86-755-86967710
邮箱:webmaster@szbl.ac.cn

系统与物理生物学研究所
zhouyq@szbl.ac.cn
AI计算生物学,分子生物学,细胞生物学
资深研究员、副所长
终身正教授
终身正教授
终身副教授
助理教授
Martin Karplus组博士后
Carol Hall组博士后
George Stell组博士后
科学家,实验室主任
化学物理博士
化学物理学士
周耀旗课题组主要围绕着RNA和蛋白质的序列、结构及功能之间的关系以及生物高分子的应用开发等几方面进行科学研究。课题组研究的特色是结构生物信息计算和现代高通量、自动进化生物技术相结合来实现对序列、结构及功能之间关系的深刻理解,从而达到生物高分子在多方面应用的这个目标,其中包括精准医疗服务的针对性药物设计和个性化生物标志物的检测。
周耀旗研究员是中国科技大学化学物理方向的学士,纽约州立大学石溪分校计算统计力学方向的博士,哈佛大学诺贝尔奖获得者Martin Karplus组计算生物的博士后。他在2000年任纽约州立大学布法罗分校助理教授,2004年升为终身副教授,在2006年成为印第安纳大学信息学院和医学院教授,2013年起是澳大利亚格里菲斯大学糖组学研究所教授, 现在是深圳湾实验室系统与物理生物学所资深研究员。
他既广又深的科研训练使他能够在美国、澳大利亚和中国工作期间带领博士生、博士后团队,领导多学科科研人员合作,在生物信息学及其在分子生物学的应用这个交叉领域多次作出独特、原创性贡献。其中包括提出“蛋白质天然态就是表面融化固体”这个新概念、一个基于统计力学方法的全原子势函数DFIRE、基于模版的蛋白质三级结构预测方法SPARKS,以及第一个把深度学习方法应用于蛋白质主链结构预测(SPIDER/SPOT-1D)以及RNA二级结构预测(SPOT-RNA),并提出利用破坏靶蛋白结构来设计不会有抗药性的小肽新药、通过高通量测序来推断RNA结构的新方法等等。曾经多次在国际蛋白质结构预测和功能预测比赛中名列前茅(蛋白质结构盲测比赛CASP6-2004服务器组第一名,CAGI-2013盲测细胞增殖率第一名,CAID-2019盲测蛋白内在无序区第一名)。
周耀旗研究员曾经荣获美国NIH、澳大利亚NHMRC和ARC、广东省重点领域研发计划项目等多个科研基金的资助,在Genome Biology, Ann. Review Biophys., PNAS, Nucleic Acids Research,Cell Metabolism,Nature Communications, Nature Machine Intelligence等国际一流杂志发表论文200多篇,根据Google Scholar,总引用1万3千多次,H-index 61,2020年引用次数1300多次。
代表性图片
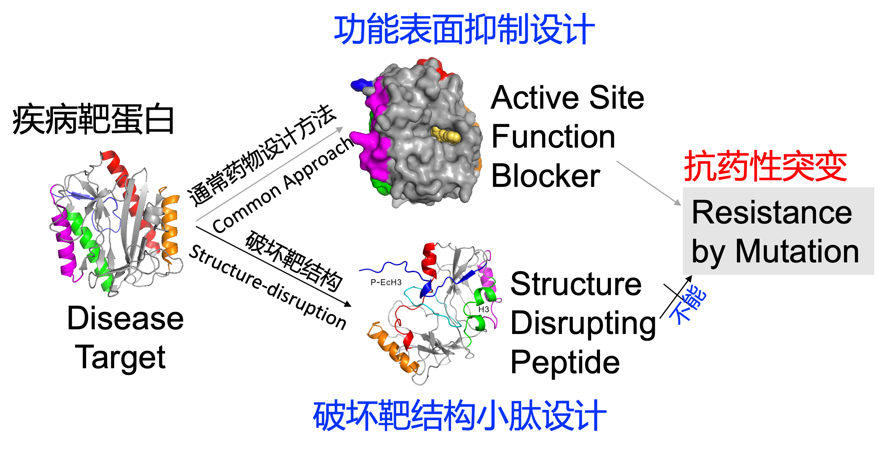
1. 目前大多数药物仅仅在药物靶标的表面上粘附,容易导致靶标突变而产生抗药性。周耀旗课题组设计的小肽能够从根本上破坏靶标的结构,从而使细菌或者癌细胞无法通过突变来产生抗药性(Zhan and Zhou et al.FASEB J., 33: 2095–2104 (2019);新闻报道(http://www.china.org.cn/world/Off_the_Wire/2018-09/28/content_64238761.htm):
2. 第一个端到端RNA二级结构预测方法大幅度地改进了预测的精度(Nature Communications10, 5407 (2019))。新闻报道(https://www.brisbanetimes.com.au/national/queensland/researchers-use-ai-deep-learning-to-predict-human-rna-structures-20191127-p53er1.html):
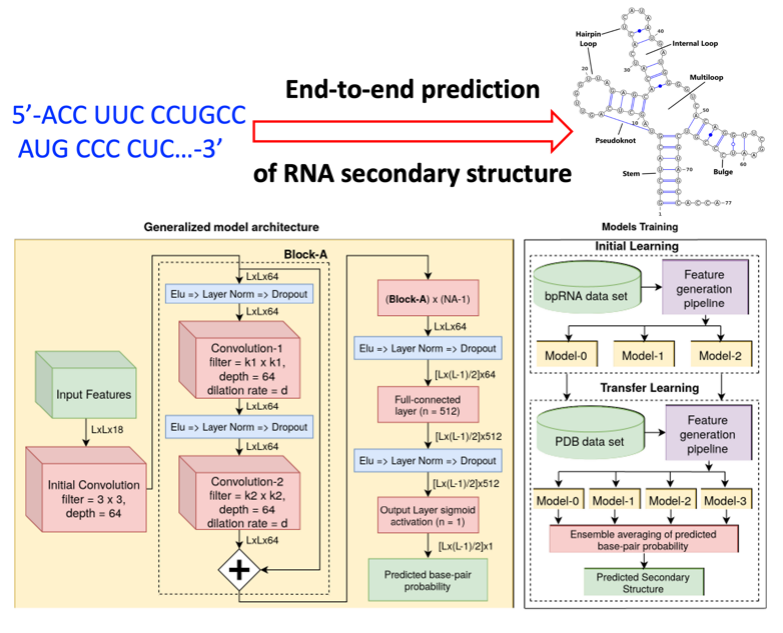
1. J. Singh, J. Hanson, K. Paliwal, and Y. Zhou, RNA secondary structure prediction using an ensemble of two-dimensional deep neural networks and transfer learning,Nature Communications10, 5407 (2019).
2. Z. Zhang, P. Xiong, T. Zhang, J. Wang, J. Zhan, and Y. Zhou, Accurate inference of the full base-pairing structure of RNA by deep mutational scanning and covariation-induced deviation of activity,Nucleic Acids Research, 48:1451-1465 (2020).
3. J. Zhan, H. Jia, E. A. Semchenko, Y. Bian, A. M. Zhou, Z. Li, Y. Yang, J. Wang, S. Sarkar, M. Totsika, H. Blanchard, F. E.-C. Jen, Q. Ye, T. Haselhorst, M. P. Jennings, K. L. Seib, and Y. Zhou, Self-derived structure-disrupting peptides targeting methionine aminopeptidase in pathogenic bacteria; a new strategy to generate antimicrobial peptides,FASEB J., 33: 2095–2104 (2019).
4. S. Xu, J. Zhan, B. Man, S. Jiang, W. Yue, S. Gao, C. Guo, H. Liu, Z. Li, J. Wang, and Y. Zhou, Real-time reliable determination of binding kinetics of DNA hybridization using a multi-channel graphene biosensor,Nature Communications8, 14902 (2017).
5. Z. Li, Y. Yang, J. Zhan, L. Dai and Y. Zhou, Energy Functions in De Novo Protein Design: Current Challenges and Future Prospects,Ann. Rev. Biophysics42, 315-335 (2013).
联系方式
地址:深圳市光明区光侨路高科创新中心
电话:+86-755-86967710
邮箱:webmaster@szbl.ac.cn
邮编:518132
Copyright © 2024 深圳湾实验室 版权所有www.szbl.ac.cn All Rights Reserved.粤ICP备19032884号 粤公网安备44031102000926号
粤公网安备44031102000926号


