
电话:+86-755-86967710
邮箱:webmaster@szbl.ac.cn

系统与物理生物学研究所
pengqin@szbl.ac.cn
力学生物学、表观遗传学、细胞生物学
特聘研究员
助理项目科学家
博士后
联合培养博士
学士
课题组主要从事染色质活细胞成像及基因表达调控机制的研究,开发针对肿瘤等疾病相关的表观遗传修饰引起的发病机制、诊断检测和治疗的成像工具,结合深圳湾实验室医工交叉的优势,从肿瘤的表观遗传调控机理和治疗等方面共同推进研究工作。
具体研究内容:为了实时“点亮”活细胞内染色质和表观遗传修饰,课题组一直从事可遗传的新型光学成像探针的开发,来解析细胞命运转化中的力-表观调控新机制,将助力肿瘤和神经退行性疾病等重大疾病发生发展的表观遗传调控新机制研究,以及新型诊疗技术和试剂的研发。主要包括以下三方面内容:
1. 开发活细胞表观遗传修饰与染色质的新型四维(4D)标记系统;
2. 在体成像与定量细胞核与染色质上所受的机械力;
3. 开发原位功能性蛋白-DNA-RNA互作组学技术,阐明衰老、肿瘤转移等过程中的力-表观调控机制,挖掘诊疗新靶点。
合成了基于全长组蛋白的荧光能量共振转移(FRET)分子探针,实现了单个活细胞水平上一种或多种组蛋白修饰的实时动态成像示踪,阐明了有丝分裂开始时H3S10ph对H3K9me3去甲基化和染色质正确重塑的调控机制。设计并开发了一款用于特异性检测RNA m6A修饰的传感器(spatiotemporal m6A imaging system, SMIS),实现了m6A修饰的RNA在活细胞中的动态可视化,揭示了m6A修饰的RNA在应激颗粒中的富集过程,发现了YTHDF2与G3BP1的相互作用促进应激颗粒的稳定性。近期开发了能同时实现活细胞内单个基因位点的示踪标记和操控的生物分子探针(Simultaneous Imaging and Manipulation with Biomolecular Assemblies,SIMBA)系统。并建立了基于定向进化技术(Directed Evolution)的高通量筛选通用平台,用于高通量构建FRET分子探针。目前还开发了一系列细胞核力学可视化测量的探针,实现了活细胞和活体上细胞核力传递的检测。在SciAdv、Nat Commun、PNAS等发表论文40余篇。
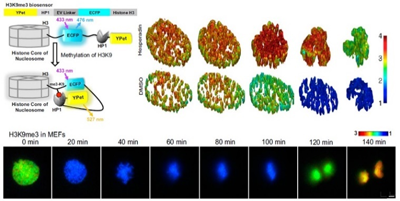
图1. 组蛋白修饰的活细胞成像
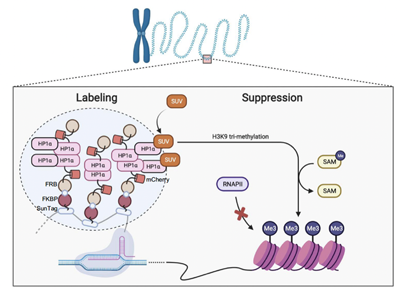
图2. 单基因位点的示踪标记和操控
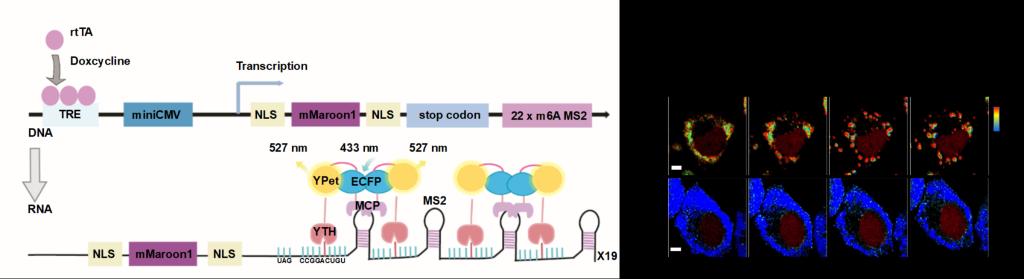
图3 RNA m6A修饰活细胞示踪系统及在应激颗粒中的应用
[1]Li Q., Liu J., Chen Y., Zhang K., Deng L., Liu H., Cheng H., Guo L., Qiu J., Wang Y.,Peng Q†. Live Cell Visualization of m6A Modification and Stress Granule Stability.Science Advances. 2024, 10(46): eadp5689.(†Corresponding author)
[2] Lin W, Tang Y, Liu J, Liu Y, Xu Y, Liu J, Liu H, Pang W, Guo X, Yao W, Zhang T†,Peng Q†, Zhou J†. Mechanical Activation of cPLA2 Impedes Fatty Acid��-Oxidation in Vein Grafts.Advanced Science, 2024: e2411559.(†Corresponding author)
[3]Li Q*, Liu H*, Du X,XieY,Chen Y, Qiu J, Gao Y†,Peng Q†.Nanobody-assisted Nano-luciferasefragmentcomplementation for in situ measurement and visualization of endogenous protein-protein interaction.Biosensors and Bioelectronics, 2024. 272: 117102.(†Corresponding author)
[4]Peng Q.*, †,Huang Z.*, Sun K., Liu Y., Yoon C., Harrison R., Schmitt D., Zhu L., Wu Y., Tasan I., Zhao H., Zhang J., Zhong S., Wang Y†. Engineering Inducible Biomolecular Assemblies for Genome Imaging and Manipulation in Living Cells.Nature Communications, 2022, 13(1): 7933. DOI: 10.1038/s41467-022-35504-x. (†Corresponding author. Featured article)
[5]Peng Q., Lu S., Shi Y., Pan Y., Limsakul P., Chernov AV., Qiu J., Chai X., Shi Y., Wang P., Ji Y., Li YJ., Strongin AY., Verkhusha VV., Izpisua Belmonte JC., Ren B., Wang Y., Chien S. †, Wang Y†. Coordinated Histone Modifications and Chromatin Reorganization in a Single Cell Revealed by FRET Biosensors.Proc Natl Acad Sci U S A. 2018, 115(50): E11681-E11690. doi: 10.1073/pnas.
联系方式
地址:深圳市光明区高科创新中心
电话:+86-755-86967710
邮箱:webmaster@szbl.ac.cn
邮编:518132
Copyright © 2025 深圳湾实验室 版权所有www.szbl.ac.cn All Rights Reserved.粤ICP备19032884号 粤公网安备44031102000926号
粤公网安备44031102000926号


