多学科交叉融合的科研时代,分子动力学(MD)模拟已成为化学、生物医药、材料科学等领域不可或缺的工具。然而,传统MD软件长期依赖国外技术,且面对大规模复杂体系模拟时,算力与效率瓶颈日益凸显。在此背景下,北京大学化学与分子工程学院高毅勤教授团队、深圳湾实验室系统与物理生物学研究所杨奕课题组、并行科技,成功完成国产分子动力学模拟引擎SPONGE的云端部署,标志着我国在自主可控的高性能计算与分子模拟领域取得重要突破。
强强联合:技术自主化与算力升级的双重突破
此次合作中,北京大学与深圳湾实验室主导SPONGE核心算法研发,其创新的GPU加速技术及AI驱动的增强采样方法(如元动力学、深度学习潜力集成等),显著提升了模拟精度与速度;并行科技则依托其云端算力平台,为SPONGE提供高性能计算资源支持,使其能够高效处理超大规模分子体系(如蛋白质-配体相互作用、纳米材料表面动力学等场景),同时支持多任务并行计算,大幅降低科研成本与时间。
这一合作通过技术开源与模块化设计(如MindSPONGE与华为MindSpore框架深度结合),为跨学科研究提供了灵活扩展的解决方案。

操作步骤一:并行云桌面登录界面

操作步骤二:点击SPONGE
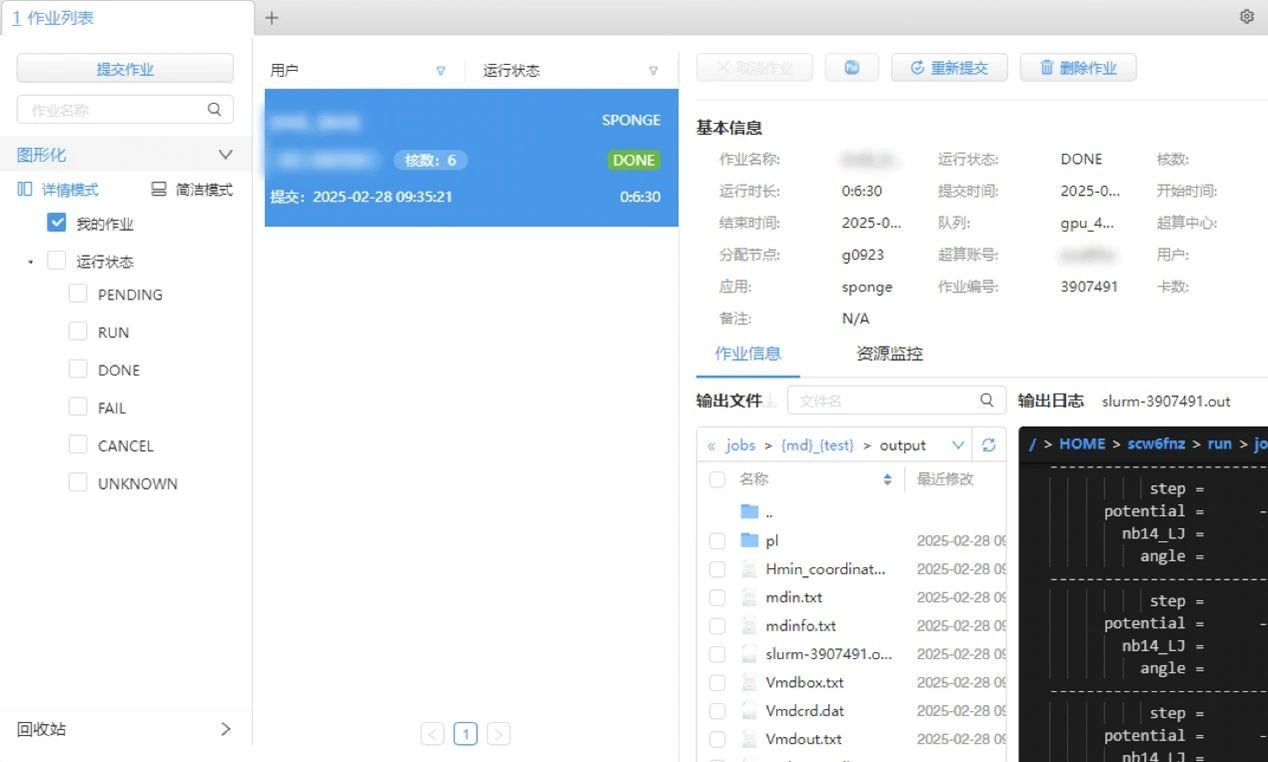
步骤三:按需在SPONGE作业列表界面进行相关作业操作
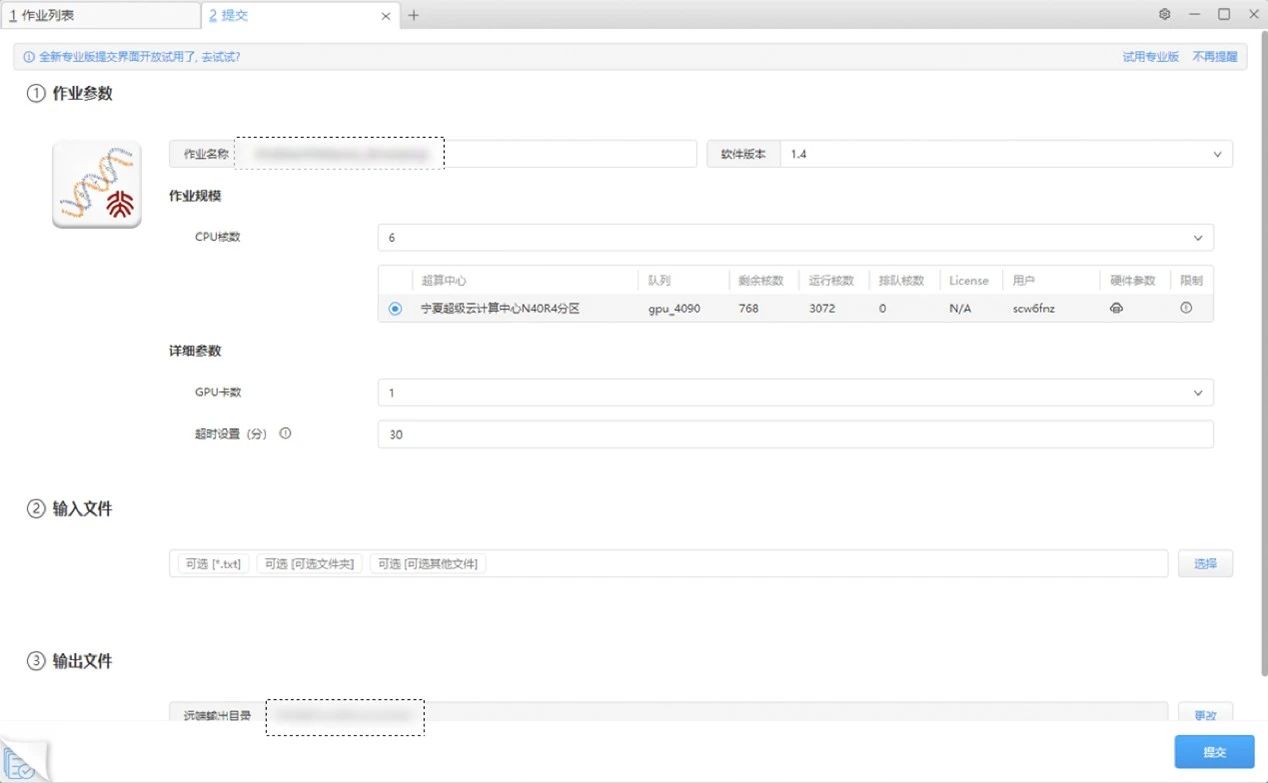
步骤四:选择作业规模并提交
用户收益:从科研到产业的全链条赋能
1. 效率跃升:SPONGE的GPU加速技术使模拟速度提升10倍以上,结合云端弹性算力,用户可快速完成传统需数周的计算任务,尤其适用于药物发现、新材料设计等时效性强的领域。
2. 精度与功能扩展:
与国外主流软件(如Amber)精度持平,且支持复杂体系(如生物大分子、特殊材料表面)的定制化模拟,满足前沿研究需求。
3. 成本优化:
云端部署无需本地高性能硬件投入,按需付费模式降低中小团队使用门槛,助力普惠科研。
4. 生态开放:
模块化设计(CudaSPONGE、MindSPONGE、Xponge)支持与深度学习、分子对接工具(如DSDP)无缝集成,推动AI for Science的深度融合。
案例实证:SPONGE的多元应用场景
-生物医药:精准模拟蛋白质构象变化,加速靶向药物筛选;
-材料科学:解析纳米材料表面动力学行为,指导高性能材料开发;
-教育领域:开源特性助力高校教学与科研训练,培养交叉学科人才。
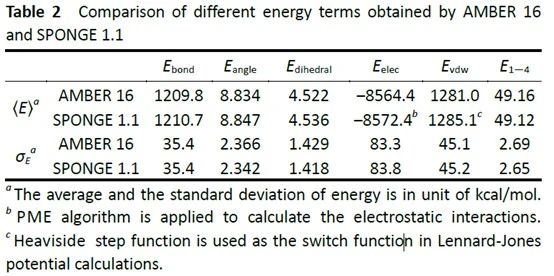
SPONGE与国外主流软件Amber计算精度上无显著差异
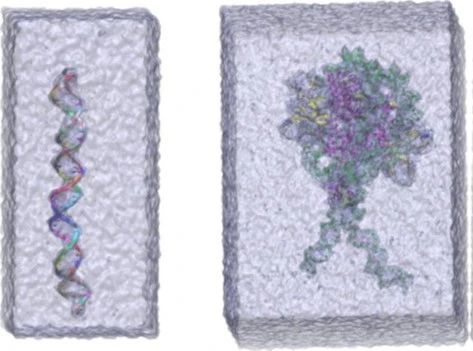
SPONGE在生物大分子体系模拟表现良好
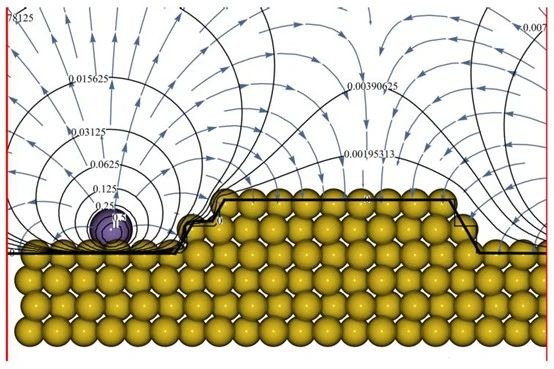
SPONGE甚至可以用于一些特殊材料体系表面信息的定制化计算
未来,随着算法与算力的持续迭代,SPONGE有望成为MD模拟领域的重要参与者,推动我国在生物医药、新材料等战略产业的核心竞争力提升。
参考文献
1. Huang Y. P. et al., *Chin. J. Chem.* 2022, DOI:10.1002/cjoc.202100456
2. Xia Y. et al., *J. Open Source Softw.* 2022, DOI:10.21105/joss.04467

 粤公网安备44031102000926号
粤公网安备44031102000926号