许多疾病(包括癌症)跟非编码RNA相关,但是我们对它们的了解甚少,因为用于理解它们功能机制的结构很难被X射线晶体衍射、核磁共振和冷冻电镜等物理方法所解析。没有机制的理解,就没有彻底解决疾病的手段,所以通过计算来预测RNA结构,大幅度提高其精度势在必行。长期以来,RNA结构的预测集中在二级结构(碱基配对),并把它用于三级结构预测的约束,但对决定整体结构的RNA主链二面角却束手无策,只能依赖于并不十分可靠的能量函数来优化,周耀旗与格里菲斯大学的Paliwal课题组合作开发的、利用深度学习来预测主链二面角的工具将改变这个状态,这个开创性成果发表在 J. Chem. Info. Modeling (2021)上。
今年,信使RNA(mRNA)在新冠疫苗研发上的巨大成功使RNA药物开发成为热点中的热点。同时,越来越多的非编码RNA被发现在许多生命过程的调控中起着极其关键的作用,并已知与越来越多的疾病相关。然而所发现的功能非编码RNA分子还不到在人体里已经发现的非编码RNA的10%,还有90%以上无法深入探索,因为它们的序列同源性弱、它们的结构我们一无所知。目前已知的实验方法对RNA结构的解析时间长、耗费大、困难多,所以提高RNA结构的计算预测精度是必由之路。
最近,谷歌AlphaFold2在第14届蛋白质结构预测的比赛(CASP14)中对大部分的蛋白实现了原子精度或接近原子精度的结构预测,从而打破了长期以来以为计算预测不可能代替实验的迷思,给RNA结构预测的前景带来了新的希望。AlphaFold2的成功主要依赖于最近几年通过深度学习方法,对蛋白质结构的主链以及蛋白质氨基酸之间的接触距离图的预测精度的大幅度改进。周耀旗课题组发展了Real-SPINE,世界上第一个用神经网络来预测蛋白质主链f和y二面角真实值的方法【1】,也发展了目前蛋白质主链二面角预测的已知最高精确度的方法之一的SPOT-1D【2】。
相比蛋白质的两个主链二面角,RNA要复杂得多。它的骨架主链是由磷酸-核糖组成,需要六个二面角(α, β, γ, ε, δ, ζ)来描述,此外由于RNA结构稳定性的关键因素是碱基对的堆叠,碱基的配对决定了磷酸-核糖主链的走向,所以连接氮碱基支链二面角c也非常重要。问题是这么多二面角,只有几百个非同源RNA结构可以作为训练集,能够预测准确吗?
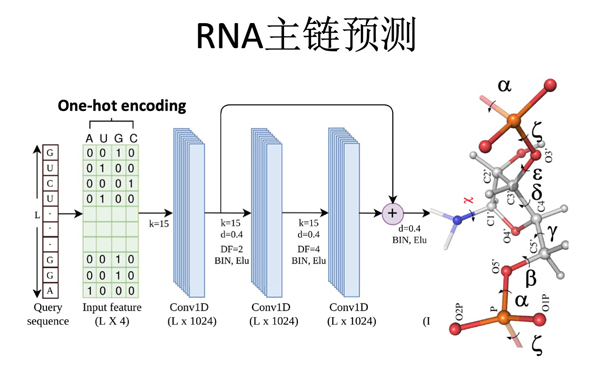
周耀旗课题组与格里菲斯大学的Paliwal课题组基于过去发展预测RNA二级结构(SPOT-RNA)【3,4】以及RNA溶剂可接触面积(RNAsnap)【5,6】的经验,利用扩张卷积神经网络(dilated convolutional neural network)来预测所有的主链及支链二面角。虽然只使用单个RNA序列作为输入信息,不同角度在三个测试集的平均绝对误差范围为14°-44°,不比单个蛋白质序列预测的φ(24°)和ψ(44°)结果差【7】,相比之下,随机预测的平均绝对误差为17°-62°。更重要的是预测角度比RNA-Puzzles里(类似于蛋白质结构预测比赛CASP的RNA结构预测比赛)最好模型的角度误差还要小,表明预测的RNA二面角将可以像预测的蛋白质二面角一样,作为RNA三级结构预测的约束。
由于RNA主链二面角是第一次被预测,这项工作受到了期刊和审稿人最严格的审视。J. Chem. Info. Modeling 共邀请六位审稿专家,提出了许多意见和建议。作者最后用更多的测试集来衡量方法的鲁棒性,以及使用RNApot和RNA puzzles结构模型库证实了预测的角度能够用来判断RNA模型是否接近天然态,而最终被接收。

资深研究员
周耀旗从今年3月起全职加入了深圳湾实验室,他是1984年中国科技大学近代化学系的学士,1990年美国纽约州立石溪大学化学物理的博士,1994-2000年北卡州立大学、哈佛大学的博士后,2000年任纽约州立布法罗大学助理教授,2004年升为终身副教授,2006年成为印第安纳大学信息学院和医学院终身正教授,2013-2021年任澳大利亚格里菲斯大学糖组学研究所正教授。他长期在结构生物信息学方面工作,曾经多次在国际蛋白质结构预测和功能预测比赛中名列前茅。到目前为止共发表论文200余篇,引用1万多次,H因子61。目前,周耀旗课题组通过计算和实验的结合,从事蛋白质/RNA的序列、结构与功能关系方面的基础研究和生物分子检测、药物开发方面的应用研究。寻找在RNA方面有计算和实验相关经验的博士后、科研助理和助理研究员。
论文标题:
RNA Backbone Torsion and Pseudotorsion Angle Prediction Using Dilated Convolutional Neural Networks
论文全文:
https://pubs.acs.org/doi/10.1021/acs.jcim.1c00153
文章来源 | 周耀旗课题组
编辑 | 鲍 啦
延伸阅读
【1】B. Xue, O. Dor, E. Faraggi and Y. Zhou, “Real value prediction of backbone torsion angles.”, Proteins 72, 427-433 (2008)。
【2】J. Hanson, K. Paliwal, T. Litfin, Y. Yang, and Y. Zhou, “Improving prediction of protein secondary structure, backbone angles, solvent accessibility, and contact numbers by using predicted contact maps and an ensemble of recurrent and residual convolutional neural networks.”, Bioinformatics, 35: 2403–2410 (2019).
【3】J. Singh, J. Hanson, K. Paliwal, and Y. Zhou, “RNA secondary structure prediction using an ensemble of two-dimensional deep neural networks and transfer learning.”, Nature Communications 10, 5407 (2019).
【4】J. Singh, K. Paliwal, T Zhang, J. Singh, T Litfin, Y. Zhou, “Improved RNA Secondary Structure and Tertiary Base-pairing Prediction Using Evolutionary Profile, Mutational Coupling and Two-dimensional Transfer Learning.”, Bioinformatics, btab165, (2021).
【5】Y. Yang, X. Li, H. Zhao, J. Zhan, J. Wang and Y. Zhou, “Genome-scale characterization of RNA tertiary structures and their functional impact by RNA solvent accessibility prediction”, RNA, 23: 14-22 (2017).
【6】A. Kumar, J. Singh, K. Paliwal, J. Singh, Y. Zhou, “Single-sequence and profile-based Prediction of RNA solvent accessibility using dilated convolution neural network.”, Bioinformatics, 36, 5169–5176(2020).
【7】R. Heffernan, K. Paliwal, J. Lyons, J. Singh, Y. Yang, Y. Zhou, Single-sequence-based prediction of protein secondary structures and solvent accessibility by deep whole-sequence learning. J. Comput. Chem. 39, 2210-2216 (2018).