Not be bound by your knowledge, as knowledge knows no bounds
李祥
Xiang David LI
粤港澳生物医学创新中心
Timeline
-
2021 至今
深圳湾实验室 资深研究员
-
2020 至今
香港大学 化学系 教授
-
2016–2020
香港大学 化学系 副教授
-
2011–2016
香港大学 化学系 助理教授
-
2008–2011
洛克菲勒大学 化学和细胞生物学 博士后
-
2003–2008
香港大学 化学系 有机化学和化学生物学 博士
-
1999–2003
复旦大学 化学系 学士
研究领域
1. 组蛋白修饰的调控和功能研究
2. 开发化学生物学方法鉴定组蛋白表观遗传修饰并表征由表观修饰介导的蛋白质相互作用
3. 开发表观修饰的“阅读器”、“擦除器”和“书写器”的化学抑制剂
4. 小分子药物蛋白靶点的鉴定和药物结合位点的表征
成果
李祥博士,香港青年科学院院士,香港大学化学系终身教授。李祥博士课题组致力于表观遗传学,化学蛋白质组学,药物化学等相关研究。实验室擅长综合利用生物化学、分子生物学、细胞生物学及生物物理学等研究手段揭示组蛋白翻译后修饰的生物学意义及其调控机制,为表观遗传学的研究提供了新思路和新方法。自独立开展工作以来,李祥博士主持多项国家自然科学基金委员会与香港研究资助局科研基金项目,在表观遗传学领域做出了突出贡献。过去十年间,多个研究成果发表于Nature Chemical Biology, Molecular Cell, Journal of the American Chemical Society, Nucleic Acids Research, Angewandte Chemie, Chemical Science, eLife, 以及Cell Chemical Biology等国际知名学术期刊,并申请美国专利2项。
荣誉
2021 香港青年科学院院士
2019 国家优秀青年科学家基金项目(港澳)
2018 中美化学与化学生物学教授协会杰出教授奖
2018 日本化学会杰出教授奖
2016 香港大学2015/16年度杰出青年研究学者
媒体报道
2.https://www.163.com/dy/article/FUI7HHM80532BT7X.html
3.https://www.163.com/dy/article/FR2CDVFG05329KGN.html
4.https://news.sina.cn/2019-11-06/detail-iicezzrr7650216.d.html
招聘信息
1. 博士后
2. 研究助理
代表论文
1. J. Lin, X. Bao, X. D. Li (corresponding author), “A tri-functional amino acid enables mapping of binding sites for posttranslational-modification-mediated protein-protein interactions”, Mol. Cell, 2021, 81, 2669.
2. Y. Jiang, G. Chen, X.-M. Li, S. Liu, G. Tian, Y. Li, X. Li, H. Li, X. D. Li (corresponding author), “Selective Targeting of AF9 YEATS Domain by Cyclopeptide Inhibitors with Preorganized Conformation” J. Am. Chem. Soc. 2020, 142, 21450.
3. Y. Jing, D. Ding, G. Tian, K. C. J. Kwan, Z. Liu, T. Ishibashi, X. D. Li (corresponding author), “Semisynthesis of site-specifically succinylated histone reveals that succinylation regulates nucleosome unwrapping rate and DNA accessibility” Nucleic Acids Res. 2020 48, 9538.
4. X. Bao, Z. Liu, W. Zhang, K. Gladysz, Y. M. E. Fung, G. Tian, Y. Xiong, J. W. H. Wong, K. W. Y. Yuen, X. D. Li (corresponding author), “Glutarylation of histone H4 lysine 91 regulates chromatin dynamics” Mol. Cell, 2019, 76, 660.
5. X. Li, Y. Wu, G. Tian, Y. Jiang, Z. Liu, X. Meng, X Bao, L Feng, H Sun, H Deng, X. D. Li (corresponding author), “Chemical proteomic profiling of bromodomains enables the wide-spectrum evaluation of bromodomain inhibitors in living cells” J. Am. Chem. Soc., 2019, 141, 11497.
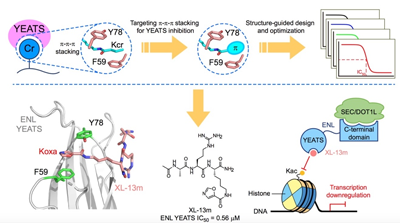
6. X. Li, X.-M. Li, Y. Jiang, Z. Liu, Y. Cui, K. Y. Fung, S. H. E. van der Beelen, G. Tian, L. Wan, X. Shi, C. D. Allis, H. Li, Y. Li, X. D. Li (corresponding author), “Structure-guided development of YEATS domain inhibitors by targeting π-π-π stacking” Nat. Chem. Biol., 2018, 14, 1140.
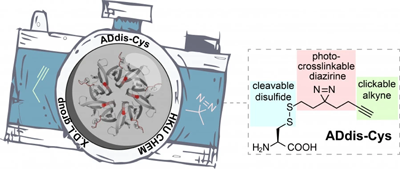
7. T. Yang, X.-M. Li, X. Bao, Y. M. E. Fung, X. D. Li (corresponding author), “Photo-lysine captures proteins that bind lysine posttranslational modifications” Nat. Chem. Biol., 2016, 12, 70.
8. Z. Liu, T. Yang, X. Li, T., Peng, H. C. Hang, X. D. Li (corresponding author), “Integrative chemical biology approaches to examine ‘erasers’ for protein lysine fatty-acylation” Angew. Chem. Int. Ed., 2015, 54, 1149.
9. X. Bao, Y. Wang, X. Li, X.-M. Li, Z. Liu, T. Yang, C. F. Wong, J. Zhang, Q. Hao, X. D. Li (corresponding author), “Identification of ‘erasers’ for lysine-crotonylated histone marks using a chemical proteomics approach” eLIFE, 2014, 3, e02999.
10. X. Bao, Q. Zhao, T. Yang, Y. M. E. Fung, X. D. Li (corresponding author), “A chemical probe for lysine malonylation” Angew. Chem. Int. Ed., 2013, 52, 4883.