内怀冰清 外涵玉润
孙坤
Kun SUN
肿瘤研究所
Timeline
-
2019 至今
深圳湾实验室 特聘研究员
-
2017-2019
香港中文大学 研究助理教授
-
2014-2017
香港中文大学 博士后
-
2014
香港中文大学 博士
研究方向
· 癌症液体活检(Cancer Liquid Biopsy)
· 肿瘤转移的分子机理与抑制方法(Cancer metastasis)
· 生物信息学(Bioinformatics/Computational Biology)
· 长链非编码RNA(Long noncoding RNA)
· 大数据挖掘、人工智能、可视化(Data mining/Augmented Intelligence/Data visualization)
更多信息(包括招聘)请访问https://hellosunking.github.io/,或添加课题组微信(sunkunlab)。
成果
孙坤博士长期从事生物信息学和基于外周血游离DNA进行癌症诊断的研究,发表SCI文章50余篇,其中(共同)第一作者或通讯作者30余篇(包括Cancer Discovery、Genome Research、Nature Communications、PNAS、EMBO Journal 等),总被引用4000余次。孙坤博士主持多项国家级科研项目,包括国家级青年人才项目、国家重点研发计划(首席科学家)等。孙坤博士的工作注重原创性和转化价值,申请了多项国际、国内专利,大部分专利均已成功转化。
孙坤课题组自成立以来以深圳湾实验室为第一或通讯作者身份已经发表了10余篇SCI论文,包括Nature Communications 2023、 Briefings in Bioinformatics 2021、Patterns 2020、Bioinformatics 2020等,详细介绍请查看https://hellosunking.github.io。
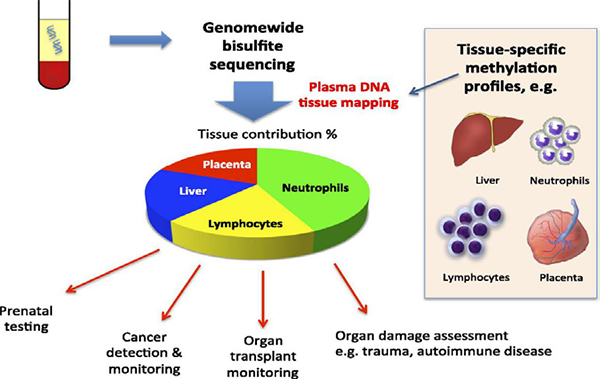
首创血浆游离 DNA 的组织来源追踪方法
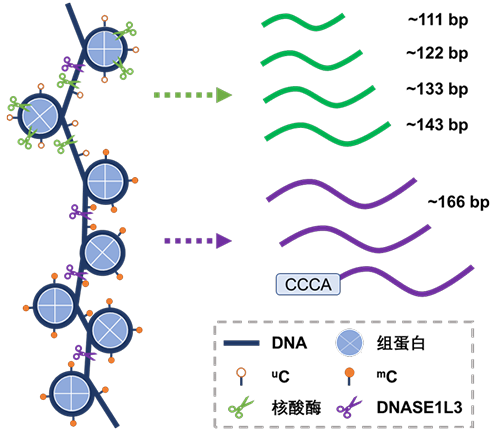
揭示游离DNA片段化模式的分子机理以及应用
荣誉
· 2021国家级青年人才项目
· 2022国家重点研发计划(首席科学家)
招聘信息
代表论文
1. An Y, Zhao X, Zhang Z, Xia Z, Yang M, Ma L, Zhao Y, Xu G, Du S, Wu X, Zhang S, Hong X, Jin X*, Sun K*. DNA methylation analysis explores the molecular basis of plasma cell-free DNA fragmentation. Nat Commun 2023 Jan 18; 14(1):287.
2. Jiang P#, Sun K#, Peng W#, Cheng SH, Ni M, Yeung PC, Heung MMS, Xie T, Shang H, Zhou Z, Chan RWY, Wong J, Wong VWS, Poon LC, Leung TY, Lam WKJ, Chan JYK, Chan HLY, Chan KCA, Chiu RWK, Lo YMD. Plasma DNA end motif profiling as a fragmentomic marker in cancer, pregnancy and transplantation. Cancer Discov 2020 May; 10(5):664-673.
3. Sun K*, Jiang P, Cheng SH, Cheng THT, Wong J, Wong VWS, Ng SSM, Ma BBY, Leung TY, Chan SL, Mok TSK, Lai PBS, Chan HLY, Sun H, Chan KCA, Chiu RWK, Lo YMD*. Orientation-aware plasma cell-free DNA fragmentation analysis in open chromatin regions informs tissue of origin. Genome Res 2019 Mar; 29(3):418-427.
4. Sun K, Jiang P, Wong AIC, Cheng YKY, Cheng SH, Zhang H, Chan KCA, Leung TY, Chiu RWK, Lo YMD. Size-tagged preferred ends in maternal plasma DNA shed light on production mechanism and show utility in noninvasive prenatal testing. Proc Natl Acad Sci U S A 2018 May 29; 115(22):E5106-E5114.
5. Jiang P#, Sun K#, Tong YK, Cheng SH, Cheng THT, Heung MMS, Wong J, Wong VWS, Chan HLY, Chan KCA, Lo YMD, Chiu RWK. Preferred end coordinates and somatic variants as signatures of circulating tumor DNA associated with hepatocellular carcinoma. Proc Natl Acad Sci U S A 2018 Nov 13; 115(46):E10925-E10933.
6. Chan KCA#, Jiang P#, Sun K#, Cheng YKY, Tong YK, Cheng SH, Wong AIC, Hudecova I, Leung TY, Chiu RWK, Lo YMD. Second generation noninvasive fetal genome analysis reveals de novo mutations, single-base parental inheritance and preferred DNA ends. Proc Natl Acad Sci U S A 2016 Dec 13; 113(50):E8159-8168.
7. Sun K#, Jiang P#, Chan KCA#, Wong J, Cheng YK, Liang RH, Chan WK, Ma ES, Chan SL, Cheng SH, Chan RW, Tong YK, Ng SS, Mong RSM, Hui DS, Leung TN, Leung TY, Lai PBS, Chiu RWK, Lo YMD. Plasma DNA tissue mapping by genomewide methylation sequencing for noninvasive prenatal, cancer and transplantation assessments. Proc Natl Acad Sci U S A 2015 Oct 6; 112(40):E5503-12.
8. Sun K*, Wang H, Sun H*. NAMS webserver: coding potential assessment and functional annotation of plant transcripts. Brief Bioinform 2021 May 20; 22(3):bbaa200.
9. Sun K*, Li L, Ma L, Zhao Y, Deng L, Wang H, Sun H. Msuite: a high-performance and versatile DNA methylation data-analysis toolkit. Patterns (N Y) 2020 Oct 15; 1(8):100127.
10. Zhou L#, Sun K#, Zhao Y#, Zhang S, Wang X, Li Y, Lu L, Chen X, Chen F, Bao X, Zhu X, Wang L, Tang LY, Esteban MA, Wang R, Jauch R, Sun H, Wang H. Linc-YY1 promotes myogenic differentiation and muscle regeneration through an interaction with the transcription factor YY1. Nat Commun 2015 Dec 11; 6:10026.
11. Lu L#, Sun K#, Chen X, Zhao Y, Wang L, Zhou L, Sun H, Wang H. Genome-wide survey by ChIP-seq reveals YY1 regulation of lincRNAs in skeletal myogenesis. EMBO J 2013 Oct 2; 32(19):2575-88.