近日,由深圳湾实验室、北京大学化学与分子工程学院高毅勤课题组与华为MindSpore团队联合开发的分子模拟库(SPONGE)发布,该软件具有高性能、模块化等特性,易于领域研发人员进行理论和算法的快速实现,推动人工智能与分子模拟的自然融合。
分子动力学模拟是用牛顿定律近似来描述微观原子和分子尺度演化的计算机模拟方法。其既可用于基础科学研究也可用于工业实际应用。在基础科学领域,分子动力学方法有助于科研学者从微观研究体系的物理化学性质。
在工业生产中,其可以利用大规模计算的能力辅助药物分子的设计和蛋白靶点的搜寻。由于模拟的时间和空间尺度限制,传统分子动力学软件的应用范围受到较大限制。科研工作者也在不断的开发新的力场模型、抽样方法以及尝试结合新兴的人工智能来进一步拓展分子动力学模拟的适用领域。
由此,新一代的分子动力学软件就需要被提上日程。其应该具有模块化的特性,能够支持科学家高效的创造和搭建出能够验证其理论模型的结构。同时,它还需要兼顾传统模拟方法的高效性,能够兼容其在传统领域上的使用。此外,为实现分子模拟+机器学习的自然融合,其还应该拥有嵌入人工智能框架的形态。SPONGE就是基于这些理念而被创造出的全新的,完全自主的分子模拟软件。
相比于之前在传统分子模拟软件上结合SITS方法进行生物分子增强抽样,SPONGE原生支持SITS并对计算流程进行优化使得其使用SITS方法模拟生物体系更加高效。针对极化体系,传统分子模拟采用结合量化计算等方式来解决电荷浮动等问题。即使采用机器学习降低计算量也会浪费大量时间在程序数据传送的问题上。而SPONGE利用模块化的特点可支持内存上直接与机器学习程序通信大大降低了整体计算时间。
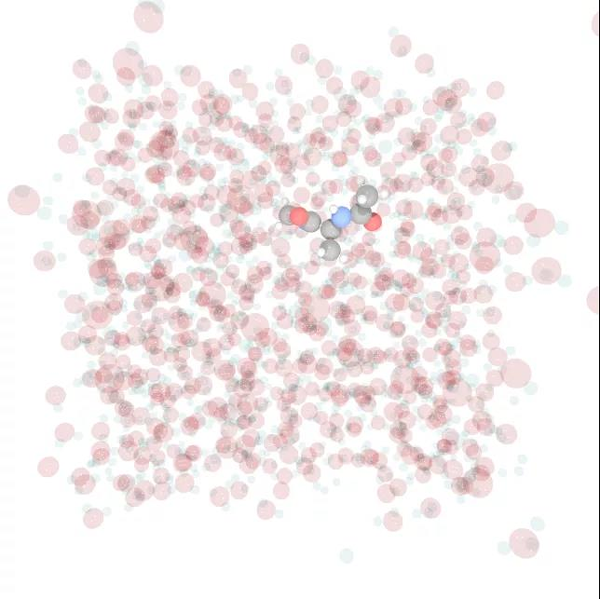
图 1:对显性溶剂中的丙氨酸二肽使用SITS方法进行增强抽样的分子动力学模拟

图 2:机器学习 + 分子模拟方法可更快更准确地模拟极化体系,图为[C1MIm]Cl 离子液体模拟
基于MindSpore自动并行、图算融合等特性,SPONGE可高效地完成传统分子模拟过程。SPONGE利用MindSpore自动微分的特性,可以将神经网络等AI方法与传统分子模拟进行结合。

SPONGE 模块化设计结构图
随MindSpore1.2版本开源的SPONGE具备以下优势:
1、全模块化分子模拟。模块化构建分子模拟算法,易于领域研发人员进行理论和算法的快速实现,并为外部开发人员贡献子模块提供友好的开源社区环境。
2、传统分子模拟与MindSpore结合的人工智能算法的全流程实现。在MindSpore中,研发人员能便利的将AI方法作用于分子模拟中。全算子化的SPONGE将与MindSpore进一步结合成为新一代端到端可微的分子模拟软件,实现人工智能与分子模拟的自然融合。
教程文档:
https://www.mindspore.cn/tutorial/training/zh-CN/r1.2/advanced_use/hpc_sponge.html
MindSpore+SPONGE 展望
近期展望
在后续的版本更新中会陆续加入已经理论验证好的MetaITS模块、有限元计算模块等功能。这些模块将帮助SPONGE能更好的从事相变和金属表面相关的模拟。同时,MindSpore版SPONGE各模块逐步支持自动微分和自动并行,对于衔接机器学习方案提供更友好的支持。
远期展望
拓展SPONGE的各种特色模块,使其能够描述大部分微观体系并同时具有较高的计算和采样效率。对特定工业需求,如药物筛选或晶型预测,将基于SPONGE衍生出完整的流程化计算方案,能够满足大规模并行计算的需求。在MindSpore框架下,SPONGE具有元优化功能,从而实现更准确和更快的力场拟合。
文章来源 | 华为云
编辑 | 鲍 啦