近日,深圳湾实验室孙坤课题组与合作者在Patterns期刊(Cell新子刊)发表了题目为“Msuite: A High-Performance and Versatile DNA Methylation Data-Analysis Toolkit”的研究论文,介绍了课题组与合作者研发的全新DNA甲基化分析软件——Msuite。该软件整合了质控、序列比对、甲基化位点检测、以及丰富的数据可视化等功能(图1)。与当前市场上的同类型软件相比,Msuite软件有更高的准确性和更低的计算资源需求。同时,除了分析传统的亚硫酸盐测序数据时通用的3碱基分析(即所有的C都转化为T)模式,Msuite软件还针对近年来新发展出的bisulfite-free甲基化分析技术提供了一种独有的4碱基分析模式。故Msuite有望成为DNA甲基化分析的一款理想化软件。
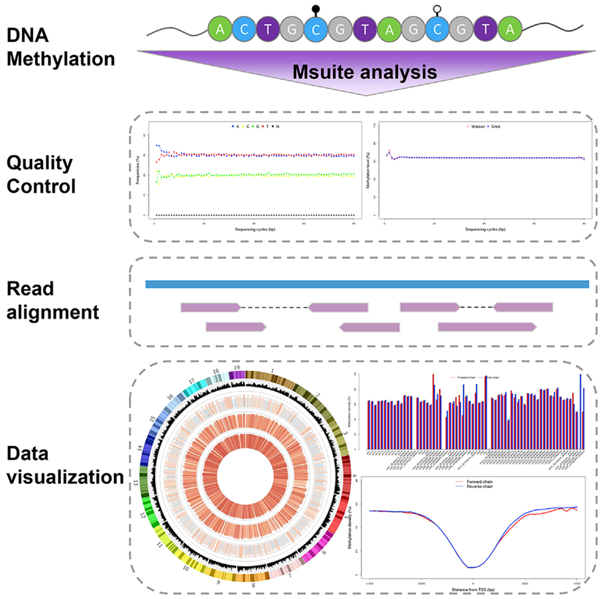
图1:Msuite的工作流程与数据可视化
DNA甲基化与分析方法
DNA甲基化是一种重要的表观遗传修饰,与许多生物调控途径有关,DNA甲基化组也是一种常见的组学研究内容。目前虽存在多种碱基级别分辨率的甲基化定量方法,如全基因组亚硫酸盐测序(whole genome bisulfite sequencing),但DNA甲基化数据的分析仍然是一项复杂且富有挑战的工作。
传统的DNA甲基化分析方法(图2),即亚硫酸盐测序,用重亚硫酸盐把未甲基化的胞嘧啶(C)转化成尿嘧啶(U),而经过甲基化修饰的胞嘧啶(C)并不受影响,仍然保持(C)碱基的状态。在后续的PCR反应中,U会被识别为胸腺嘧啶(T);因此根据互补配对原则,之后的PCR产物中,未甲基化的C都变成了T而甲基化的C保持不变。在哺乳动物中,被甲基化修饰的C大部分出现在CpG二核苷酸结构(胞嘧啶C后面紧接着鸟嘌呤G)当中,该结构只占了全部C的很少一部分(人的基因组中约5%)。因此,亚硫酸处理会使得DNA中占绝大部分的未甲基化的C转变为T,从而导致GC含量大幅下降,非常影响测序的准确性。
近年来,研究人员开发出了多种新兴的甲基化分析方法。这些新方法的共同特色是:不采用亚硫酸盐处理,而是使用其他转化酶,经过一系列的化学反应,将甲基化的C转化成T,而未甲基化的C保持不变,因此可统称为bisulfite-free方法。使用bisulfite-free方法处理的DNA序列只有很小一部分C发生了改变,因此保持了较高的序列复杂性,并对上述GC偏差的问题有了很好的改善。此外,bisulfite-free方法也具有更低的DNA降解率,适用范围更广泛。
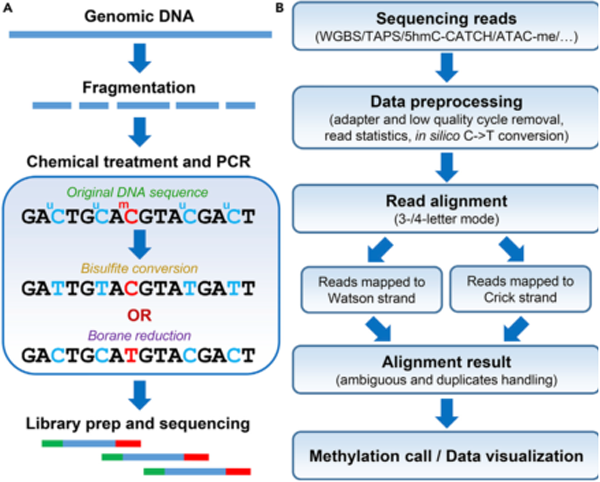
图2:DNA甲基化研究方法,左侧为实验技术,右侧为Msuite的分析流程
无论是亚硫酸盐测序还是bisulfite-free方法,都会将一部分C转化为T,转化后的DNA并不能直接比对到基因组。目前的主流分析方法都是为亚硫酸盐测序所设计,大多使用了3碱基匹配模式(把参考基因组和测序序列的C都转成T),但是该模式并没有发挥出bisulfite-free技术的序列复杂度的优势。还有一些软件采用通配符匹配法(即忽略C和T之间的错配),速度较慢,匹配率也有所欠缺。此外,当前软件的集成度较低,需要用户自己做质控和下游分析。
为了应对上述问题,孙坤课题组开发了Msuite软件,集成了质控、序列比对、甲基化位点检测和丰富的数据可视化等功能,旨在为当前大多数DNA甲基化分析提供一款全功能的软件包(图2)。Msuite包含了一种全新的4碱基匹配算法,专门为新兴的bisulfite-free方法的数据进行设计和优化。和当前的同类型工具相比,Msuite有更高的精准度、匹配率,同时对于计算资源的需求也更少。因此,Msuite有望成为DNA甲基化数据分析的理想软件,尤其可以促进新兴的bisulfite-free方法的普及。
Msuite的特色和优势
研究人员将Msuite和目前的主流软件进行了详细比较,结果表明,Msuite在功能和兼容性上均优于其他软件(表1),而且更加易于使用。同时,Msuite在比对率、准确度、运算时间和内存需求上都具有明显优势(表2)。
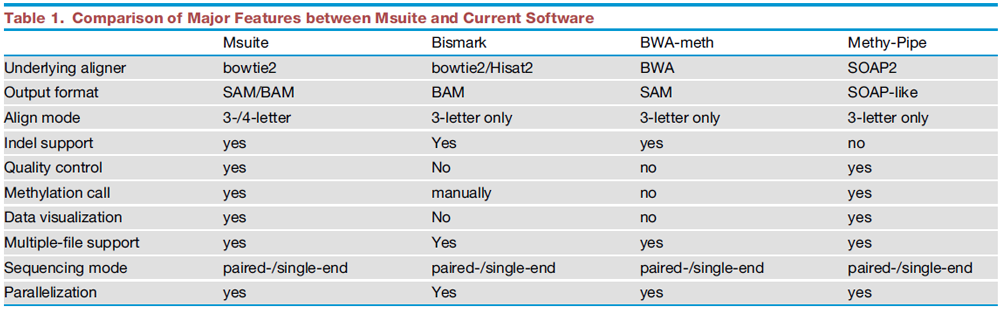
表1:Msuite与主流软件的功能特性比较

表2:Msuite与主流软件的性能对比
此外,Msuite提供详细的质控信息和丰富的数据可视化功能(图3)——以网页形式提供数据分析中的关键参数,供用户评估数据的质量;输出的分析结果可使用UCSC Genome Browser、IGV或者Circos等软件进行可视化;其附带的Mviewer软件可提供一种具有特色的数据可视化方案(图3C),尤其适于展示基因型相关的甲基化模式。
深圳湾实验室博士后李丽诗、研究助理马丽,中山大学赵喻教授,深圳湾实验室分子生理研究所特聘研究员邓麟,香港中文大学王华婷教授、孙昊教授共同参与完成本项研究。
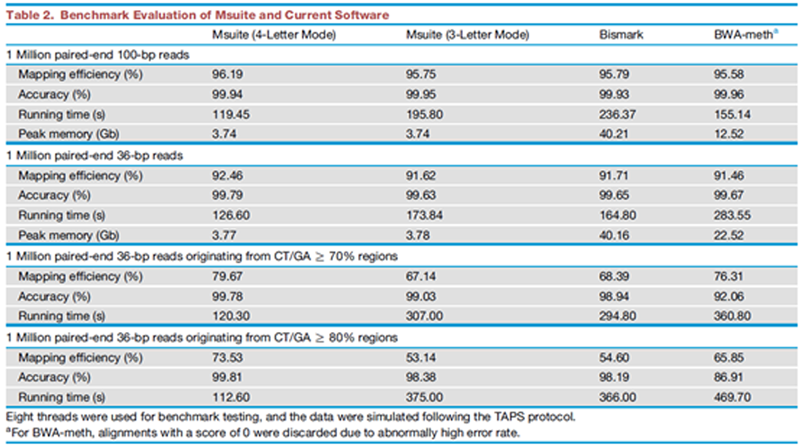
图3:Msuite提供丰富的数据可视化功能
课题组介绍:
孙坤 特聘研究员
孙坤博士2014年在香港中文大学获得博士学位,并于2019年全职加入深圳湾实验室,任特聘研究员。孙坤博士长期从事生物信息学和基于外周血游离DNA的液态活检(无创产前测试和癌症诊断)方向的研究,拥有近10年的生物医学大数据分析经验,发表SCI文章40余篇,其中(共同)第一作者或通讯作者20余篇(包括Cancer Discovery、PNAS、Genome Research、EMBO Journal、Nature Communications、Briefings in Bioinformatics等),总被引用2300余次。孙坤博士的工作注重原创性和转化价值,曾参与申请多项国际专利,其中一些已经转让给GRAIL、Illumina等多家液态活检领域内的知名公司。
孙坤博士课题组主要研究方向包括但不仅限于:(1) 生物信息学软件开发;(2) 大型数据集信息挖掘;(3) 高准确度、低成本的癌症生物标记物筛选;(4) 单细胞技术在分子诊断方向的应用探索。
健康大数据时代,生物信息学在生物医学的很多领域都扮演着重要的角色,孙坤博士课题组诚邀有志之士加入!
论文标题:Msuite: A High-Performance and Versatile DNA Methylation Data-Analysis Toolkit
论文全文:https://www.cell.com/patterns/fulltext/S2666-3899(20)30168-9
Msuite下载地址:https://github.com/hellosunking/Msuite
撰稿 | 孙坤课题组
编辑 | 白 白、远 山